1. 서론
병리학 및 제약 분야에서 질병의 종류 및 약물 처리에 따른 세포의 형상 및 운동에 대한 관찰이 이루어져 왔다. 기존의 대표적인 세포 영상 촬영 방법으로는 경우 세포의 대략적인 형태를 관찰하는 광학 현미경이나 형광 물질을 사용해 세포 내 특정 단백질의 위치 및 역할을 확인하는 형광 현미경이 있으며, 금속염을 사용해 세포를 염색하는 전자 현미경 방식도 널리 사용되어왔다. 하지만 기존의 방법은 살아있는 세포의 활동을 관찰하는 방식이 형광 물질로 염색이 가능한 단백질에 국한되어 있었으며, 광학 현미경으로 촬영된 영상의 경우, 세포의 3차원 형상을 정량적으로 표현하기에 부족한 한계가 존재했다. 이를 해소하기 위한 방법으로 최근 레이저 간섭형 현미경 기법에 기존의 의료 영상의 단층 촬영법 아이디어를 적용한 광 회절 단층 촬영법 (Optical Diffraction Tomogarphy, ODT)이 개발되었다 [1].
광회절 단층 촬영법을 통해 얻어진 세포 영상은 단백질 밀도에 따라 달라지는 굴절률을 정량적인 수치로 나타낸 3차원 볼륨으로 구성되어 있으며, 세포의 전체 형상에 대해 별도의 염색 없이 이미징 할 수 있다는 특징을 가지고 있다. 이러한 새로운 세포 영상 모달리티는 단층 촬영 기법을 차용했다는 점에서 CT나 MRI와 같은 의료 영상과 유사한 데이터 형태를 지니고 있으며, 이를 기존의 세포 영상 가시화 결과와 유사하게 나타낼 수 있는 프레임워크에 대한 필요성이 대두되고 있다.
본 논문에서는 의료 영상 가시화 라이브러리를 활용해 ODT 영상 내의 세포 데이터를 2차원 단면 및 3차원 볼륨으로 가시화 하고 영상 내의 특정 밝기 범위를 원하는 색의 3차원 볼륨 가시화 결과로 나타낼 수 있는 복합 가시화 기법을 제시하고자 한다.
2. 관련연구
세포의 3차원 가시화에 대한 기존 연구들은 주로 형광 현미경과 전자 현미경을 통해 얻어진 영상에 대해 이루어져왔다. 오랜 기간에 걸쳐 발전되어 정립된 촬영 기법들인 만큼 최근 들어서는 가시화 방법 자체에 대한 새로운 접근 보다는 특정 병변이나 세포 활동에 특화된 분석 인터페이스나 대용량 세포 데이터의 실시간 가시화에 대한 연구가 이루어져 왔다 [2,3].
형광 현미경과 같이 염색을 바탕으로 한 라벨 기반 영상은 세포 전체의 구조를 보는 것이 아닌 특정 단백질, 세포 소기관 단위 혹은 세포 단위로만 관찰이 가능하기 때문에 세포 전체의 조도 값을 구할 수 없으며 따라서 특정 조도 값을 강조하는 볼륨 가시화 기법을 활용했다. 또한 전자 현미경의 경우 금속염을 활용한 염색 과정에서 세포의 생체 활동이 정지되고, 세포 표면만을 촬영할 수 있기 때문에 표면의 형상을 입체적으로 나타내기 위한 깊이 정보를 포함하는 알파-값 블렌딩 (alpha-blending)을 주로 활용했다. 하지만 ODT 영상의 경우, 영상 내 복셀 (voxel)의 밝기 차이가 세포 내부 구조를 구분할 수 있는 유일한 척도가 된다는 점에서 기존의 라벨 기반 영상들과 차이가 있으며, 사용자가 해당 영상에서 특정 구조를 임의로 분리해 내고 해당 구조의 특징을 강조할 수 있는 가시화 기법이 필요하다.
본 논문에서는 가시화 라이브러리인 VTK (Visualization ToolKit)와 영상 라이브러리인 ITK (Insight Segmentation and Registration Toolkit) 등의 Open-source 라이브러리의 기능들을 바탕으로 DICOM, NIFTI와 같은 의료 영상 데이터 가시화 및 정량화에 특화된 라이브러리인 MITK (Medical Imaging Interaction Toolkit) 기반 소프트웨어 상에서 ODT영상을 다룰 수 있는 프레임워크를 구축했으며 [4,5] MITK의 영상 가시화 모듈을 세포 데이터의 구조 및 특징에 맞게 발전시킨 고급 가시화 기법을 개발했다.
3. ODT 영상을 활용한 3 차원 생세포 가시화 기법
본 장에서는 ODT 세포 영상 데이터를 의료 영상 가시화 툴에서 활용하기 위한 데이터 변환 및 사용된 볼륨 가시화 기법, 그리고 자연스러운 세포 형상의 표현을 위한 가시화 결과 방법에 대해 다룬다.
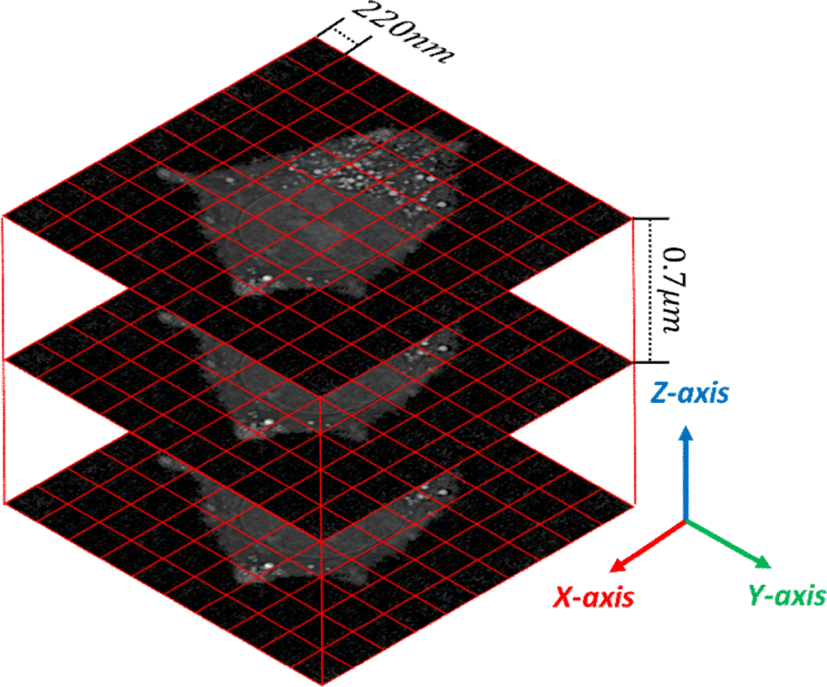
의료 영상의 경우 복셀을 구성하는 밝기 값의 범위의 오더가 103 (CT의 경우에는 약 -2000에서 2000사이) 인 반면 ODT 세포 영상에서의 복셀 값은 굴절률 (Refractive Index, RI) 로 약 1.3 에서 1.5 와 같이 상대적으로 작은 스칼라 값 범위를 가진다. 따라서 세포 영상에 대해 의료 영상과 유사한 볼륨 렌더링 기법을 적용하기 위하여 영상을 불러오는 과정에서 복셀 값이 적절한 범위를 가지도록 리스케일 (re-scale)하는 데이터 변환 작업을 수행해 볼륨 가시화 기법으로는 가장 널리 사용되는 광선 투사법 (ray-casting)을 적용했다. 하지만 방사선, 자기장 등을 피사체를 둘러싸는 원형의 궤적을 따라 360도로 회전시키며 투사해 영상을 획득하는 의료 영상과 달리 ODT 영상의 경우 세포를 통과하여 CCD로 도달하는 투사광의 각도를 반사경 조작을 통해 반구 범위 안에서 조절함으로 인해 X축과 Y축에 비해 Z-축 해상도가 많이 낮다는 특징을 가지고 있는데 이는 해상도 와 관련해 발생하는 볼륨 렌더링 상의 문제를 야기 할 수 있다는 문제가 있다.
Figure 1은 현미경으로 획득된 ODT 영상의 모식도로 X, Y축은 220 nm 간격의 해상도를 가지고 있지만 Z 축은 0.7 μm 로 상대적으로 3분의 1 이하의 낮은 해상도를 지님을 알 수 있다. 해상도에 따라 특정 위치마다 복셀 정보가 없는 빈 공간을 가진 영상 데이터에 반해 실제 세포는 연속적인 특징을 지니고 있기 때문에 복셀 들 간의 보간을 통해 연속적인 3차원 형상을 표현해야 한다. 광선 투사법은 광선이 진행하는 방향에서 정해진 간격 마다 복셀 값을 샘플링 해 광선이 만나는 물체 표면의 정보를 바탕으로 최종적인 픽셀 값을 결정한다. 하지만 가시화 하고자 하는 굴절률 범위가 좁아질 경우, 복셀 정보가 존재하지 않는 빈 공간을 보간하기 위한 참조 복셀의 정보가 적어지면서 Figure 2(b) 와 같은 나뭇결 잔상이 생기는 결함 (wood-grain artifact)이 발생하며, Z축 방향에서는 보간을 위한 복셀 정보 부족으로 인해 세포 형상이 제대로 표현되지 않는 문제가 발생한다.

나뭇결 결함의 이상적인 해결 방법으로는 샘플링 거리를 복셀 간격 보다 줄이는 오버 샘플링(over-sampling)을 진행하는 방법이 있지만 샘플링 횟수 증가로 인한 렌더링 속도 저하의 문제가 있다.
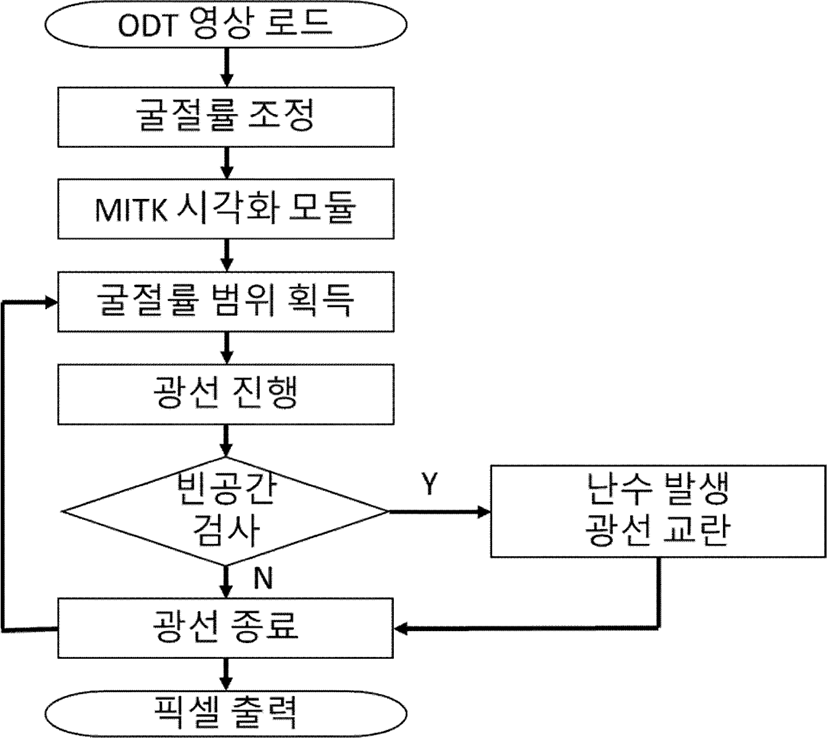
이에 가시화 속도를 유지하며 나뭇결 결함을 해소하기 위해 교란 샘플링을 (jittered sampling) 적용했으며, 광선이 출발하는 카메라의 위치에서부터 광선 상의 샘플까지의 거리 값을 난수 (random number)를 바탕으로 교란하여 샘플링을 비일관적으로 진행하는 방법을 통해 ODT 데이터의 빈 공간 때문에 발생하는 주기적인 무늬가 눈에 띄지 않게 했다. 세포 데이터의 3차원 가시화를 위한 전체 과정은 Fgirue 3과 같다.
Figure 4는 빈 공간 교란 기법 통해 복셀 정보 없이 보간 (interpolation)을 통해 표현되는 영역에서 물결무늬가 나타나지 않는 개선된 가시화 방법으로 표현된 적혈구 영상이다. 사각형 내부의 확대 영상에서 보간 된 빈 공간에서 일정한 패턴의 잡음인 나뭇결 결함이 난수의 형태에 따라 흩어져 부드럽게 나타나는 모습을 확인할 수 있다.

4. 세포 영상 가상 염색 기법
본 연구에서는 경계가 모호한 ODT 영상 데이터로부터 효율적으로 세포 내 구조를 분리해 내기 위해 시행착오 (trial-and-error) 과정을 거쳐 최적의 결과를 도출하도록 유도하는 가상 염색 기법 제안한다. 3장에서 언급한 볼륨 렌더링 기법을 바탕으로 볼륨 내 복셀이 지니고 있는 고유한 값과 색상 (color) 및 투명도 (opacity) 를 매핑해 볼륨을 구성하는 구조를 시각적으로 분리 할 수 있으며, 원본 데이터 및 지정된 영역의 단면에 대해 2차원 오버레이 (2D overlay) 가시화를 제공하는 것을 통해 사용자로부터 정의된 전이 함수에 따른 실제 복셀 영역을 원본 영상의 전체 세포 구조와 함께 확인 할 수 있도록 영상 단면상의 영역으로 표현했다 (Figure 5).
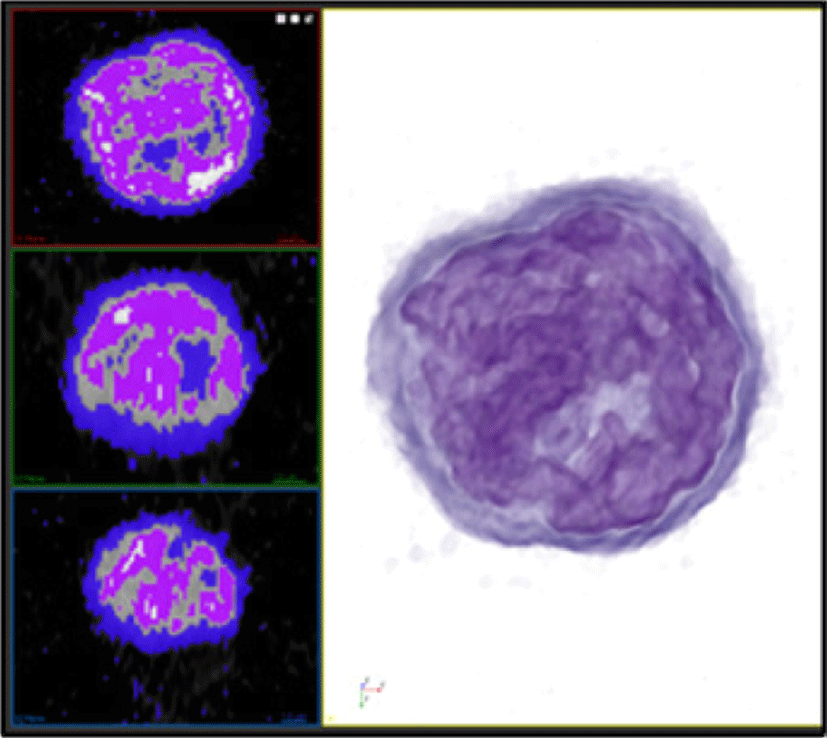
가상 염색은 비가역적 작업인 실제 염색과 비교했을 때, 반복 수행을 바탕으로 다양한 가시화 결과를 도출 할 수 있다는 장점을 가지지만, 실제 형광 현미경의 주 역할인 화학 물질을 바탕으로 세포 내 활동에 직접적으로 관련된 단백질의 위치를 특정하는 기능을 재현하기 어렵다는 한계가 있다.
이후의 장에서는 세포 내 구조의 복잡성과 ODT 영상 외 실제 염색 영상과 같은 추가적인 모달리티에 유연하게 대응하기 위한 시각화 기능 및 인터페이스에 대해 설명하도록 한다.
5. 세포 영상 복합 가시화 기법
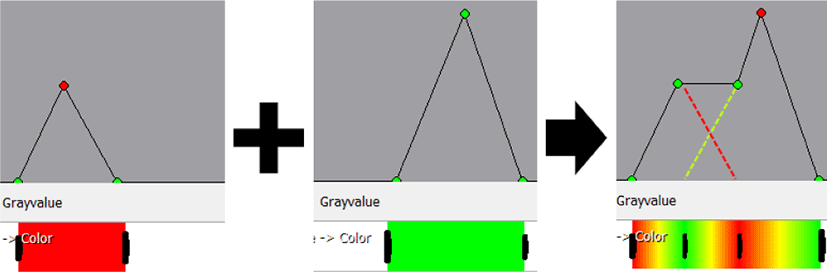
고차원 전이 함수는 볼륨 데이터의 시각화 과정에서 보다 복잡한 색상 매핑을 가능하게 하여 저차원 전이 함수로는 정의 할 수 없는 형상을 도출할 수 있지만, 원본 영상을 기준으로 하지 않기 때문에 사용자가 의도한 범위를 결정하기 어렵다는 단점이 존재 한다 [6].
제안된 시각화 프레임워크에서는 ODT 영상 촬영 기법을 통해 획득한 세포 데이터로부터 다양한 형상을 정의 할 수 있도록 기존의 복셀의 굴절률 (Figure 6 (a))을 기준으로 하는 1차원 전이 함수 대신 영상의 기울기 (Gradient Magnitude, GM) 정보 (Figure 6 (b))를 동시에 활용 하는 2차원 전이 함수 개념을 활용했다. 사용자는 지정된 굴절률의 범위와 굴절률의 기울기의 정도 범위 내에 들어가는 복셀 들의 집합에 대해 특정 색상과 투명도를 결정한다. 기울기 정보는 굴절률이 급변할수록 높은 수치를 지니는 영상의 에지 (edge)로 이해할 수 있어, 사용자가 어렵지 않게 지정된 전이 함수의 의미를 이해 할 수 있다.
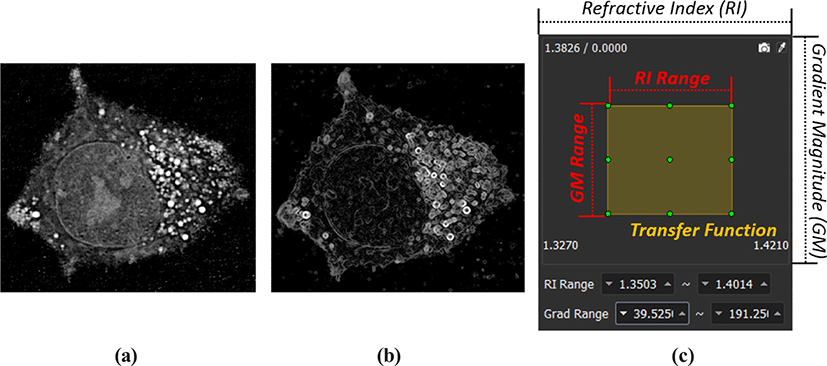
추가적으로, 2차원 전이 함수의 두 가지 정보를 직관적으로 다루기 위해 가상의 캔버스 상에 사각형 형태의 핸들을 생성하고 조작해, 세포 데이터의 볼륨 렌더링 결과를 얻을 수 있는 가상 염색 인터페이스를 구축했으며, Figure 6 (c)에서의 사각형 정보가 굴절률 및 기울기의 범위를 나타낸다. 사각형의 색상 및 투명도가 전이 함수를 통해 매핑 될 복셀 들의 성질 (property)을 나타내며, 가상 캔버스에서 가로 축은 ODT 영상 전체의 굴절률 범위를, 세로축은 기울기 범위를 나타낸다.
한편, ODT 영상에서 동일한 굴절률을 가지는 복셀이라도 복셀 집합의 위치와 형상에 따라 세포 내에서 서로 다른 구조를 의미할 수 있기 때문에 하나의 볼륨에 대해 두 개 이상의 전이 함수가 존재할 경우 함수의 정보를 혼합하지 않고 독립된 개체로서 유지 시킬 필요가 있다.
예를 들어 Figure 7의 빨간 원 안의 구조는 내핵이며 나머지 구조는 세포 소기관을 구성하는 복셀들이 가시화 된 결과이다.
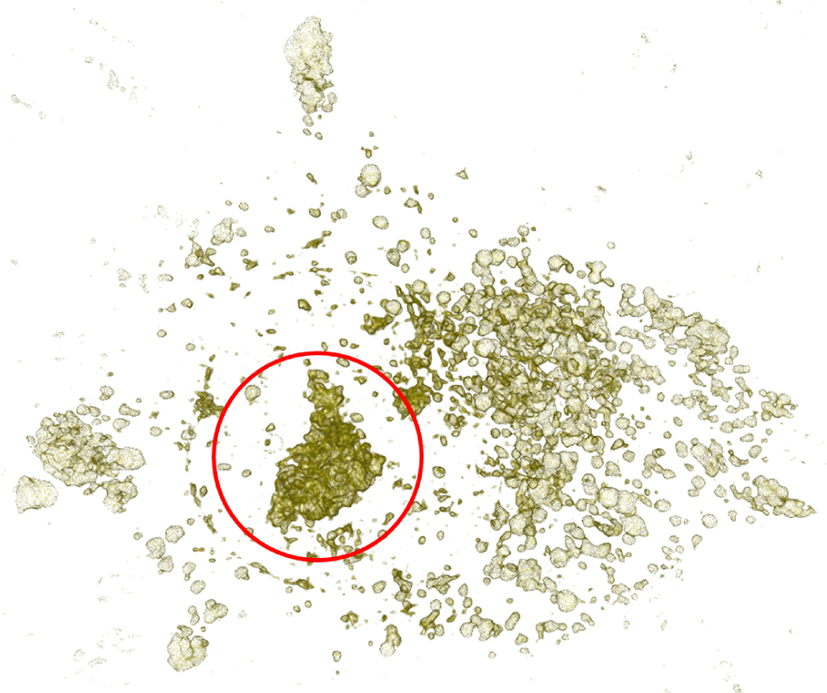
Figure 8은 MITK 라이브러리에서 제공하는 가시화 툴을 활용해 재현한 다중 전이 함수 보간의 예를 보여주는데, 중복되는 복셀의 밝기 범위에 대해 색상과 투명도의 보간이 이루어져 새로운 전이 함수가 정의됨을 확인 할 수 있다.
자동으로 생성 된 전이 함수를 바탕으로 새로운 성질에 매핑 된 새로운 복셀 영역이 나타나지만 단일 영상의 정보만을 참조 하는 것만으로는 다양한 종류와 형상의 세포에 대해 일관성 있는 보간 결과를 생성하는 전이 함수 융합 기법을 정의하기 어렵다는 점에서 세포 형상 가시화에 있어 전이 함수의 보간은 적합하지 않다.
가상 염색을 통한 세포 구조 가시화에서 다중 전이 함수에 대해 고유의 범위와 성질로 대표되는 복셀 영역을 구분해 가시화 하고, 나아가 ODT 영상이 가지는 세포의 전체 구조에 대한 표현력과 형광 현미경 영상의 세포내 활동에 대한 해석 기능의 장점을 동시에 활용하기 위해 서로 다른 특성을 가진 세포 가시화 결과를 통일된 인터페이스 상에서 나타낼 필요가 있다. 광선 투사법을 활용해 세포 내 다중 구조를 구분하여 가시화 하는 작업에서 동일한 공간상에서 상이한 색상, 투명도를 가지는 볼륨의 영역을 볼륨 겹침 이라고 정의 할 수 있으며 크게 두 가지로 구분 할 수 있는데, 첫 번째는 하나의 볼륨에서 동일한 복셀이 여러 개의 전이 함수를 참조할 경우이며, 두 번째는 광선이 두 개 이상의 볼륨을 지나갈 때이다. 본 연구에서는 언급된 두 가지 볼륨 겹침 현상을 각각의 전의 함수와 서로 다른 데이터로 정의되는 복셀 집합을 분리해 가시화 한 뒤 하나의 공간에 합쳐서 표현하는 레이어드 렌더링 (layered rendering) 기법을 제안했다 (Figure 9).

각각의 데이터는 독립되어 가시화 되고 결과는 레이어 (layer)로 저장되고, 레이어들을 겹쳐 최종적으로 하나의 3차원 가시화 결과로 나타나도록 했다. 이때, 앞에 있는 레이어가 뒤의 레이어들을 가려 아래쪽 레이어 일수록 지니고 있는 가시화 결과가 어두워지기 때문에 각 레이어가 지니고 있는 정보의 특성과 중요도에 따라 겹침의 순서를 일관성 있게 결정해주기 위해 레이어 오더링 (layer ordering) 알고리즘을 제안했다 (Figure 10).
레이어의 순서를 정하는 과정은 전이 함수가 참조하는 영상의 종류에 따라 별도의 집합으로 분리하여 각각의 집합에 대해 전이 함수의 범위를 기준으로 정렬하는 순서로 이루어진다. 정렬이 완료되면 상대적으로 밝은 특성을 지닌 레이어들이 위쪽으로 이동하며 영역 정보가 중요한 의미를 가지는 형광 단층 영상이 형상 정보에서 의미를 찾을 수 있는 ODT 영상의 위쪽으로 이동하게 된다.
Figure 11은 ODT 데이터 가시화 예를 통해 레이어드 렌더링 기법을 설명하고 있다. Figure 11 (a)에서 진핵 세포 중 하나인 뉴런의 전체 형상과 세포 소기관을 가시화하기 위한 세 가지 전이 함수가 포함하는 복셀 영역을 확인할 수 있다. 전이 함수를 제작하는 과정에서 안쪽의 가장 높은 굴절률을 가진 소기관부터 바깥쪽의 전체 형상 순서로 정의했기 때문에 차례로 레이어를 쌓은 렌더링 결과는 Figure 11 (b)와 같으며 외부 구조인 세포벽이 안쪽 기관들을 가려 관찰이 어려운 것을 확인 할 수 있다. Figure 11 (c)에서는 레이어 오더링 알고리즘을 적용해 레이어가 가진 전이 함수의 특성에 따라 정렬된 렌더링 결과를 확인 할 수 있다.
6. 가시화 기법 적용 사례
가시화 기법 중 볼륨 렌더링은 MITK의 시각화 모듈인 VTK 의 GPURayCastMapper를 활용해 구현되었으며, 세가지 채널 (빨강, 초록, 파랑)의 형광 단층 영상들을 합쳐 하나의 RGB 영상를 만드는 방법으로 ODT 영상과 함께 2차원 단면을 생성 생성했다. 또한 전이 함수를 다루기 위한 Canvas의 경우 GUI 라이브러리인 QT를 활용해 제작 되었다.
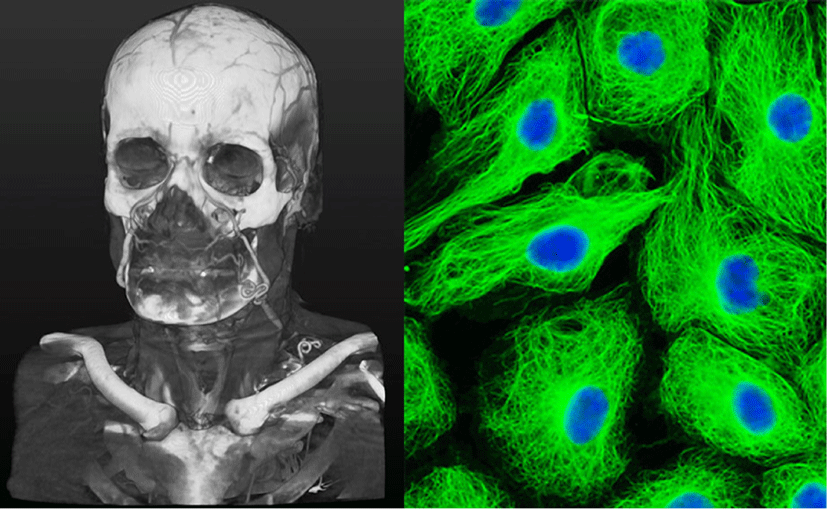
Figure 12는 일반적인 의료영상의 가시화와 세포 염색 영상의 가시화의 차이점에 대해 보여주고 있다. 의료 영상 가시화에서는 대표적인 인체 장기의 표면 정보와 주변 장기를 나타내는 전략을 지니고 있는 반면 세포 영상의 경우 염색된 영역이 지니고 있는 고유의 밝기 및 구조를 강조함을 알 수 있다.
기존의 의료 영상 가시화 기법을 그대로 세포 영상에 적용 할 경우 세포의 구조만을 표현하는 반면 2차원 전이 함수를 활용할 경우 세포의 내부 구조뿐만 아니라 영상의 에지 정보를 강조하는 것을 통해 두 개 이상의 세포 내부 구조를 보다 명확하게 전달 할 수 있다 (Figure 13). 제안된 시각화 프레임워크의 특징은 특히 세포가 지닌 얇은 막 형태의 구조를 표현하는데 특화되어 있으며, Figure 14의 세포막과 같이 세포 전체의 형상을 구성하는 세포벽이나, 세포의 핵막과 같은 얇은 구조를 표현하는데 용이하다.
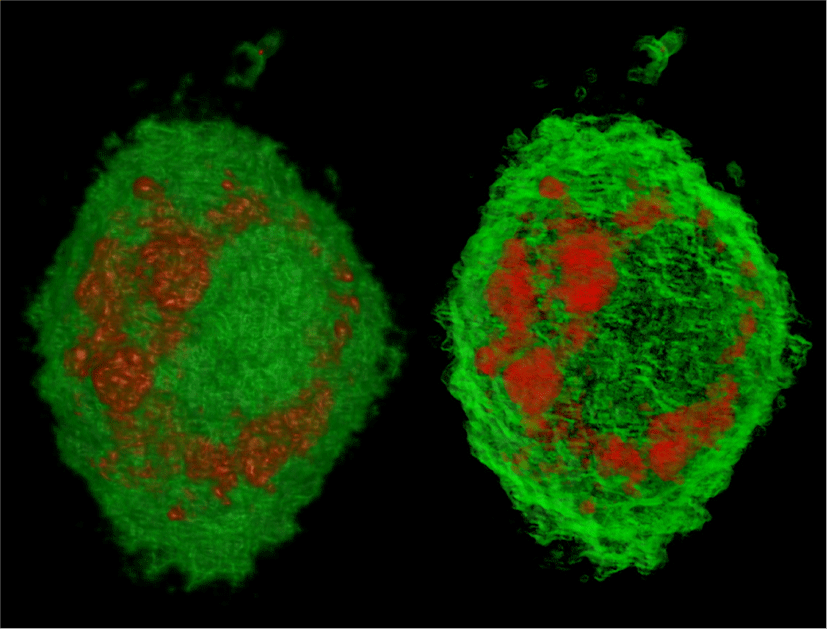
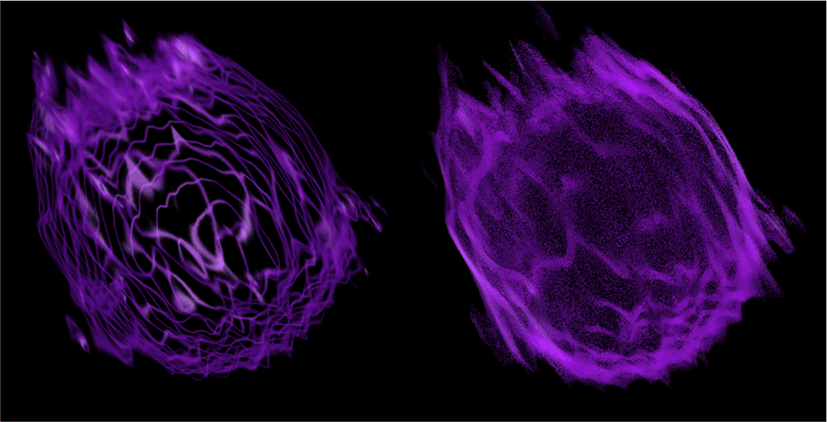
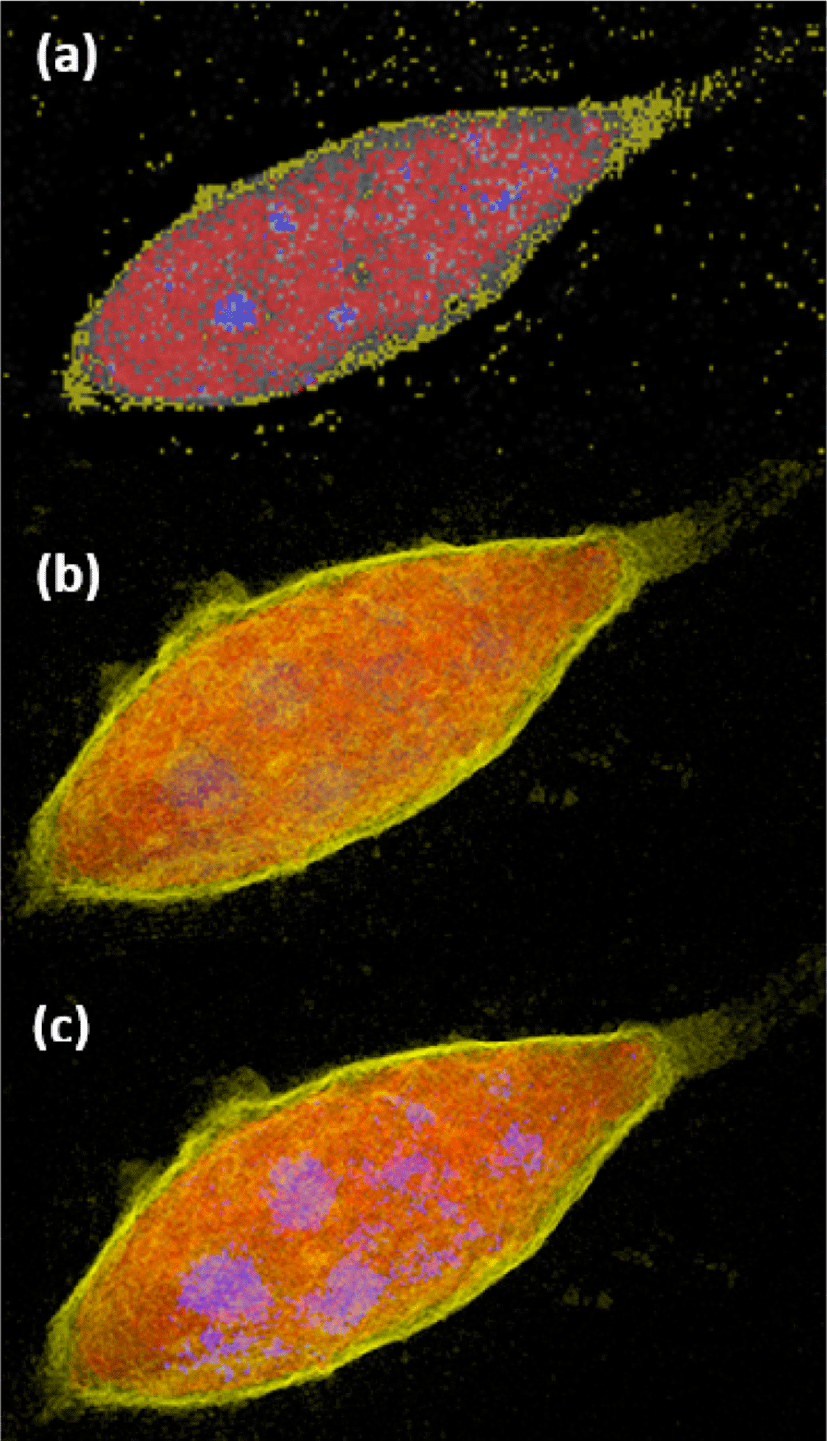
Figure 15에서 파란색으로 나타난 부분은 염색 영상의 파랑 채널로부터 정의된 핵막을 나타내며 나머지 부분은 ODT 영상을 활용한 가상 염색으로 얻어진 세포의 전반적인 구조이다. 핵막 내부에서 빨간색으로 가시화 된 구조는 세포핵의 유전 물질과 관련된 단백질이며, 핵막 주변에 형성되어 있는 구조에 대해서는 소포체와 같은 세포핵 주변 기관이라는 추가적인 분석이 가능하다.
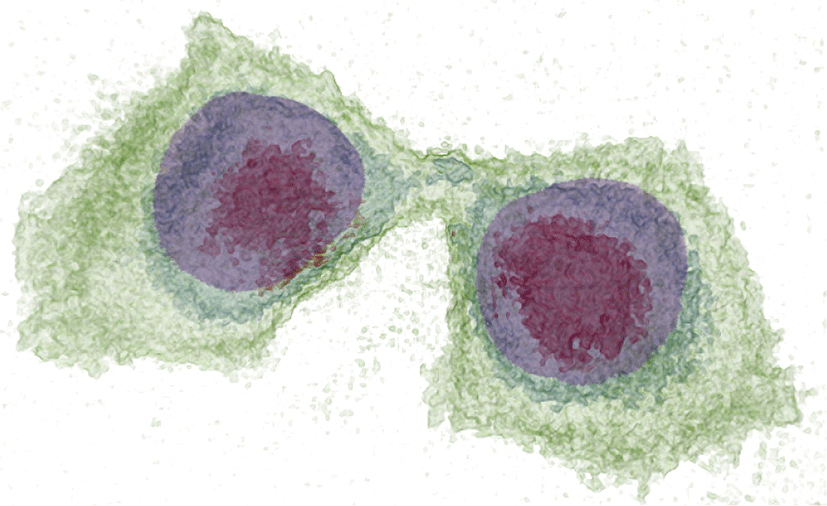
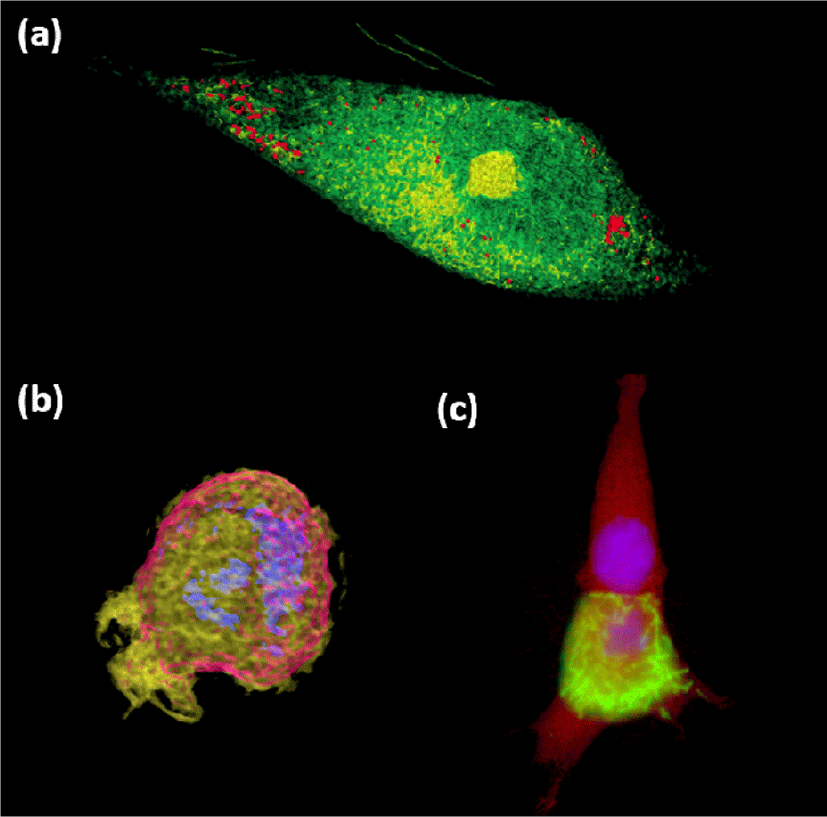
본 연구에서는 제안된 시각화 프레임워크를 다양한 ODT 및 형광 영상에 적용해 보았으며, figure 15는 그 대표적인 예시들이다. 특히 figure 15 (b) 에서 분홍색으로 나타난 세포벽이 망가지며 노란색의 세포질이 외부로 유출되는 현상을 관찰 할 수 있으며, figure 15 (c) 에서는 ODT 데이터로부터 세포 전체의 형상 (빨간색) 및 형광 영상 데이터로부터 염색된 세포핵 (파란색)과 세포피층 (cell cortex, 초록색)을 동시에 표현하고 있다.
6. 결론 및 향후 연구
본 연구를 통해 기존의 의료 영상 가시화 툴을 활용해 세포의 형상 정보를 비침습적으로 촬영 할 수 있는 ODT 영상에 대한 3차원 가시화 기법을 제안했으며, 다양한 종류의 세포에 대해 의미 있는 가시화 결과를 확인하고 그 의미를 추론 해볼 수 있었다. 향후 연구로는 전이 함수의 보간 방식과 레이어 정렬 방식을 단순히 굴절율 범위를 바탕으로 진행하는 것 대신 사용자 입력을 학습해 세포의 종류와 특정 병변에 특화된 가시화 결과를 도출 할 수 있도록 가시화 기법을 발전시키고자 한다. 제안된 세포 영상 가시화 프레임워크는 시각적 피드백을 바탕으로 한 정량화 가이드 제공 및 정량화 결과 개선 인터페이스의 초석이 될 것으로 기대한다.