1. 서론
생성 모델(generative model)은 비지도 학습을 통해 실제 데이터의 분포를 학습하여 유사한 데이터를 생성하는 모델 이다. 생성 모델은 분포를 근사하는 방법에 따라 변분 오 토인코더(Variational Autoencoder, VAE)[1], 생성적적대신경 망(Generative Adversarial Network, GAN)[2] 등 다양하게 연구되었다. 의료영상 분야의 다양한 응용 연구에서도 생 성 모델을 활용한 연구가 활발히 진행되고 있다. 예를 들 어, 의료영상을 사용한 분류(classification) 및 분할 (segmentation) 응용 연구에서는 생성모델을 통해 훈련 데 이터를 증강하고, 적은 양의 데이터와 클래스 간 불균형 문제[3]를 해결할 수 있는 연구가 발표되었다. 이렇듯 생성 모델을 활용한 연구가 많아지면서 생성된 영상의 정확한 평가에 대한 중요성도 높아지고 있다.
그러나 대부분의 의료 영상 증강 관련 연구에서는 생성 영상 품질에 대한 평가를 위해 생성한 영상을 바탕으로 학 습한 분류 및 분할 모델의 성능 개선 측면에서 데이터 증 강의 효과를 분석하는 정도에 그치거나[4, 5], 전문가의 시 각적 튜링 테스트를 수행하거나[5, 6, 7, 8, 9], 생성 영상의 특징 분포를 시각화하는 수준에 머물러 있다[10, 11, 12].
시각적 튜링 테스트를 통한 분석 방법은 전문가에게 실 제 영상과 생성 영상을 보여주고 해당 영상이 실제 영상인 지 생성 영상인지 구분함에 따라 점수를 부여하는 방식이 다. 평가 시 사용하는 생성 영상은 생성 모델로부터 임의 로 샘플링 된 영상을 사용한다. 그러나 이러한 방식은 전 문가마다 주관적인 평가 기준이 있을 수 있고 이로 인해 점수를 객관적으로 정량화하기 어렵다는 한계점이 있다[13]. 생성 영상의 특징 벡터 시각화를 통한 분석 방법은 합 성곱층(convolution layer)에서 데이터의 고차원 특징들을 추출한 후, t-SNE (t-Distributed Stochastic Neighbor Embedding) 알고리즘을 이용하여 이 특징들을 저차원으로 임베딩하고 2차원의 도표에서 시각적으로 비교하는 방법이 다. 이 방법은 생성 영상의 특징들 간의 분포가 잘 분포되 어 있는지를 시각적으로 확인할 수 있다는 장점이 있지만, 특징 벡터 시각화 방법은 정량화된 수치로 비교 및 분석을 하지 못하는 한계점이 있다.
이러한 한계점들을 극복하기 위해 생성 영상의 품질과 다양성 측면에서 생성 영상을 정량적으로 평가하는 방법이 있다. 품질은 생성 영상이 실제 영상을 얼마나 정확하고 유사하게 묘사하는지 판단하는 기준(criteria)이며, 다양성은 생성 영상이 실제 영상의 전체 분포를 얼마나 포함하는지 판단하는 기준이 된다[13, 14]. 의료 생성 영상에 대한 정 량적 분석 방법을 사용한 기존 연구에서는 IS(Inception Score) 평가 방식[15]을 사용하여 생성한 영상의 품질과 다 양성을 반영하여 평가하거나, FID(Fréchet Inception Score) 평가방식[9, 16]을 사용하여 생성 영상과 실제 영상 간의 유사도를 평가했다. 이와 같은 평가 방식은 생성 영상에 대해 정량화하여 수치적인 분석은 가능하나, 영상의 품질 과 다양성 측면을 분리하여 고려하지 못하고 하나의 결과 값으로 평가하는 1차원 점수 기반 평가 방법이라는 한계가 있다. 최근 컴퓨터비전 분야에서는 품질과 다양성이라는 두 가지 요소를 분리하여 평가하는 새로운 평가방법이 제 안되었으나[14], 이를 의료 영상에서 사용하여 평가한 연구 는 아직 활발하지 않다.
본 연구에서는 생성한 의료 영상을 품질과 다양성 측면 에서 평가를 하기 위해, 컴퓨터 비전 분야에서 평가를 위 해 사용하는 정량적 평가 방법들을 의료 영상에 적용하고 자 한다. 이를 위해 간단한 합성곱층의 GAN 구조를 가진 DCGAN(Deep Convolutional Generative Adversarial Networks)[17]과 성능 개선 기법이 다양하게 적용된 PGGAN(Progressive Growing Adversarial Networks)[18] 모 델을 이용하여 흉부 CT 영상을 학습하고, 두 생성 모델의 생성 영상을 정량적 분석 방법을 통해 평가하고 결과를 분 석하였다. 또한, 기존의 정량적 분석 방법 중 IS, FID와 같 은 1차원 점수 기반 평가 방법만으로는 해석할 수 없었던 생성 영상들의 품질과 다양성을 Precision 및 Recall 계열 의 2차원 점수 기반 평가 방법을 이용하여 분석하고 의료 영상에서의 해당 평가방법들의 특징 및 한계점을 분석하고 자 한다.
2. 생성영상 성능 평가 방법
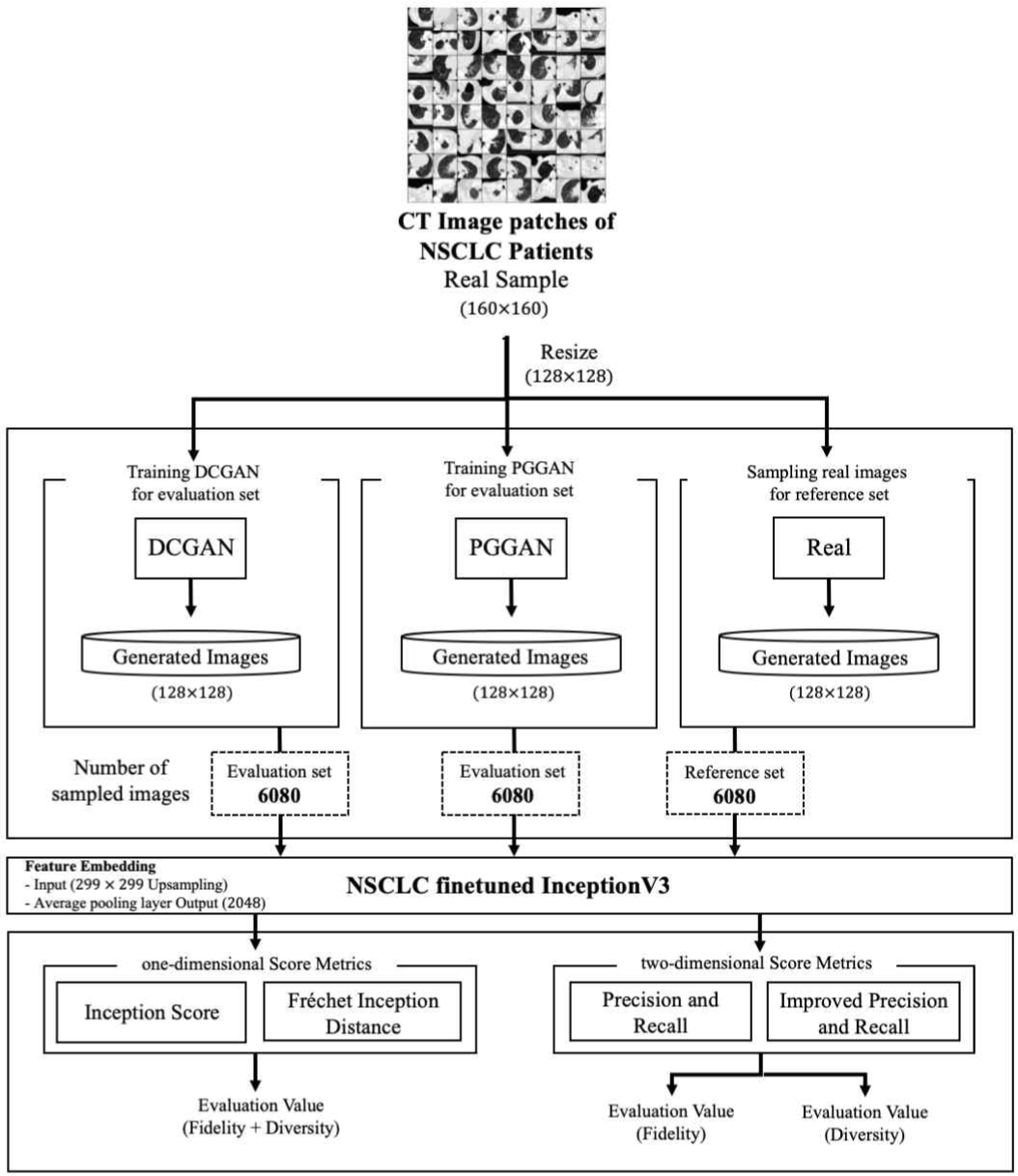
그림 1은 본 연구의 의료 영상 생성 및 정량적 평가 방 법에 대한 개요도를 나타낸다. 먼저, 종양을 중심으로 크롭 된 160 × 160 크기의 흉부 CT 영상을 128 × 128 크기로 리사이징한 후 생성적적대신경망인 DCGAN과 PGGAN 모 델에 각각 학습시킨다. 둘째, 학습 후 128 × 128 크기로 생 성된 영상에서 랜덤하게 샘플링하여 생성 영상에 대한 평 가데이터를 만들고, 원 영상에서 랜덤하게 샘플링하여 기 준데이터를 만든다. 셋째, ImageNet으로 사전 학습된 InceptionV3 모델을 학습 데이터로 파인 튜닝 후, 튜닝된 모델의 특징 공간으로 임베딩하고 1차원 및 2차원 점수 기 반 평가 방법으로 성능을 평가한다.
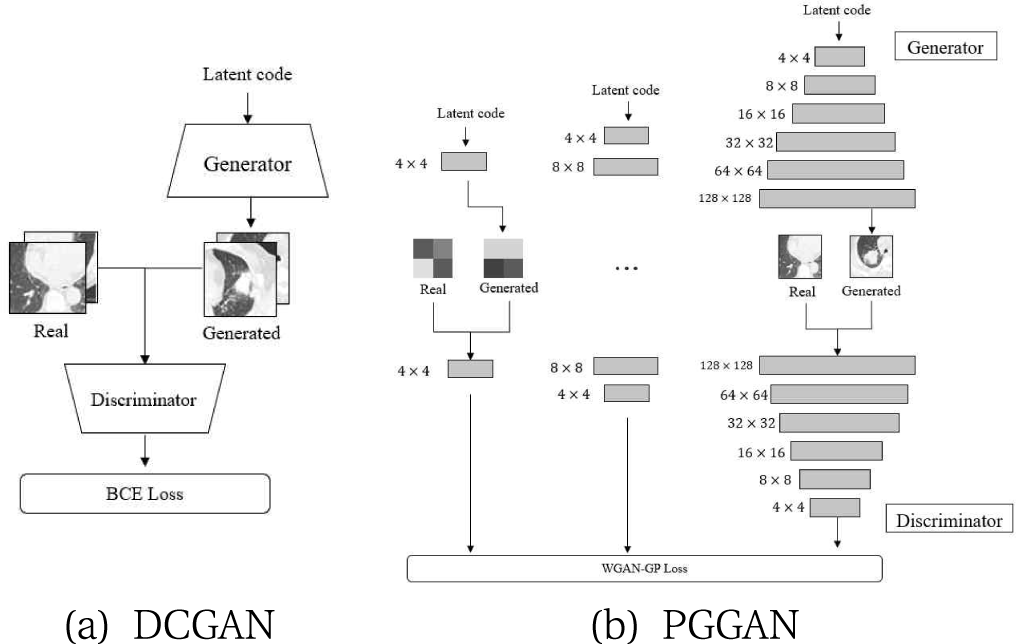
본 논문에서는 의료 영상을 생성하기 위해 GAN 기반 모델인 DCGAN과 PGGAN을 사용하여 학습한다. 그림 2 는 DCGAN과 PGGAN의 학습 방식에 대한 그림이다.
DCGAN은 합성곱층을 사용한 GAN 기반 모델로, 생성 자(Generator)는 transposed convolution을 사용하여 업 샘플링하며 마지막 층을 제외한 모든 층에서 배치 정규 화(batch normalization)를 사용한다. 각 층마다 활성화 함수로 ReLU 함수를 사용하고 마지막 층에서는 Tanh 함수를 사용한다. 판별자(Discriminator)는 풀링층을 사 용하는 대신 strided convolution을 사용하며 마지막 층 을 제외한 모든 층에서 배치 정규화를 사용한다. 각 층 마다 활성화 함수로 Leaky ReLU 함수를 사용하고, 마지 막 층에서는 Tanh 함수를 사용한다. DCGAN은 손실함 수로 이진 교차 엔트로피를 사용하며 정규분포를 따르 는 100차원의 랜덤 변수를 입력으로 받는다.
PGGAN은 점진적인 층을 쌓아서 영상을 생성하는 GAN 기반 모델로, 영상의 전반적인 형태를 먼저 학습 후 세부적인 특징들을 학습하며 생성자와 판별자를 학 습한다. 영상의 크기를 저해상도인 4 × 4에서 고해상도 인 1024 × 1024까지 점진적으로 층을 쌓아가며 안정적 인 학습을 진행하고, 생성되는 영상의 다양성이 개선되 는 장점을 갖는다. PGGAN은 학습을 위해 생성자에 3 × 3 합성곱층 후 픽셀 간 정규화 기법(pixelwise normalization)을 사용하고, 미니배치 표준편차 기법을 판별자에 적용하여 다양성에 대한 학습효과를 증진한다. 또한, 학습률 균등화 기법을 사용하여 판별자와 생성자 의 학습 속도를 동일하게 보장하고, 손실함수로 WGAN-GP[19]를 사용하며 정규분포를 따르는 512차원의 랜덤 변수를 입력으로 받는다.
DCGAN 모델은 현재 64 × 64 크기의 영상을 생성할 수 있지만 고해상도의 영상을 생성할 때는 학습이 불안 정하다는 한계점이 있다. 반면, PGGAN 모델은 1024 × 1024 영상까지 생성할 수 있으며 고해상도의 영 상을 안정적으로 생성할 수 있는 장점이 있다.
1차원 점수(one-dimensional score)기반의 생성 영상 성 능평가 방법은 단일의 결과점수를 갖는 평가방법으로, 품 질과 다양성의 정도를 합쳐 점수를 부여하는 방법이다.
IS[20]는 ImageNet 데이터셋으로 사전 학습된 InceptionV3 모델의 분류기를 이용하여 식 1과 같이 조건 부확률 p(y|x)과 주변 확률 p(y)의 확률분포 차이를 KL-Divergence를 통해 구한 점수 값이다. 생성 영상의 품 질이 좋으려면 조건부 확률 분포의 엔트로피(entropy)는 낮 아야 하며, 생성 영상의 다양성이 높으려면 주변 확률분포 의 분산이 커야 하므로 주변 확률 p(y)의 엔트로피는 높 아야 한다. 따라서 이상적인 IS 점수는 두 확률분포 간의 차이를 계산했을 때 값이 클수록 생성 영상이 잘 만들어졌 다고 평가한다.
이 때, x를 생성 영상, y를 클래스 레이블, p(y|x)는 조건 부확률로 영상 데이터를 InceptionV3 모델에 입력해 얻을 수 있는 클래스 y의 확률이다.
FID[21]는 InceptionV3 모델의 특정 층을 사용하여 데이 터를 특징 공간으로 임베딩 후 식 2와 같이 생성 영상과 실제 영상간의 평균과 공분산을 이용하여 두 데이터의 분 포간의 차이를 계산하는 평가방식이다. 두 데이터의 분포 는 다변량 정규분포라고 가정하며, FID의 계산 결과가 작 을수록 두 데이터는 유사하다고 판단한다.
이 때, μx 와 μy는 각각 실제 영상과 생성 영상으로부터 추출한 특징 분포의 평균을 의미하며 와 는 실제 영 상과 생성 영상으로부터 추출한 특징 분포의 공분산을 의 미 한다.
IS는 생성 영상의 다양성과 품질에 대한 상관관계를 점 수에서 잘 보여주고 있으나, 생성모델이 근사한 영상 분포 하나만을 가지고 평가하기 때문에 실제 영상과의 정확한 비교가 불가능하다. 또한, InceptionV3 모델의 클래스에 없 는 영상을 평가할 경우, 클래스에 영향을 받으며 과적합 (overfitting)과 모드붕괴 현상(mode collapse)을 잡아내지 못한다는 한계점이 있다[13]. 반면, FID는 생성된 영상을 실제 영상과 비교하기 때문에 IS 보다 정확한 비교 분석이 가능하다. 또한, 공분산 계산 비교를 통해 영상의 다양성에 대한 변화를 인지할 수 있기 때문에 모드붕괴 현상을 잘 잡아낼 수 있다[13]. 그러나 IS와 FID은 1차원 점수지표이 기 때문에 영상 분포의 다양성과 품질을 분리하여 제시하 지 못한다[22].
2차원 점수(two-dimensional score)기반의 생성 영상 성능평가 방법은 두 개의 결과점수를 갖는 평가방법으 로, 품질과 다양성에 대한 점수를 각각 부여하는 방법이 다.
Precision 및 Recall[14]은 품질과 다양성을 분리하여 평가하기 위해 제안된 평가지표이다. Precision은 생성 영상 중 실제 영상 샘플 분포에 속한 비율을 의미하며, 생성 영상이 얼마나 정밀하게 실제 영상 샘플을 묘사하 였는가에 대한 정밀도를 의미한다. Recall은 실제 영상 중 생성 영상 샘플 분포에 속한 비율을 의미하며, 실제 영상이 얼마나 많이 생성 영상을 통해 재현되었는가를 나타내는 재현율을 의미한다. 따라서 Precision이 높을수 록 생성 영상의 품질이 좋다고 판단하고, Recall이 높을 수록 생성 영상의 묘사의 다양성이 높다고 판단한다. Precision 및 Recall의 계산은 InceptionV3 네트워크의 중간층을 이용하여 생성 영상과 실제 영상의 특징을 추 출하고, K-평균 군집화(K-means clustering)를 통해 영상 들의 군집을 나눈다. 이와 같이 군집 안에 속한 생성 영 상과 실제 영상을 기반으로 Precision과 Recall 값을 구 한다. 이 후에 식 3과 같이 조화 평균에 가중치(β)를 부 과하는 Fβ Score 계산을 사용하여 최종 Precision과 Recall 값을 계산한다.
이 때, β > 1은 Recall에 가중치를 주고, β < 1은 Precision에 가중치를 주어 Precision과 Recall의 최종 두 수치값을 표현한다. Precision 및 Recall은 상대적인 밀 도에 의존하여 계산하기 때문에 모드 붕괴나 단절 (truncation)에 의해 생성 영상들이 밀집화 되어 있는 현 상을 제대로 설명하지 못한다는 한계점이 있다.
개선된 Precision 및 Recall[23]은 InceptionV3 네트워 크의 중간 계층을 이용하여 생성 영상과 실제 영상의 특징을 추출한다. 이 후 실제 영상과 생성 영상의 영역 (manifold)를 K-최근접 이웃(K-nearest neighbor) 방식을 통해 구(hypersphere)로 근사한다. 식 4는 개선된 Precision을 구하는 식으로, 실제 데이터의 K번째로 가 까운 실제 영상의 영역에 생성 데이터가 존재하는지 유 무를 이진값으로 구하여 평균낸다. 식 5는 개선된 Recall을 구하는 식으로, 생성 데이터의 K번째로 가까운 생성 데이터의 영역에 실제 데이터가 존재하는지 유무 를 이진값들을 구하여 평균 낸다. 식 6은 특징벡터간의 거리를 비교하기 위해 정의된 식이며, K번째 특징벡터 보다 가까운 벡터가 존재할 경우 1, 존재하지 않을 경우 0을 출력하는 함수식이다. 따라서 생성 영상 중 실제 영상 영역에 생성된 생성 영상이 많이 위치할수록 Precision이 높으며, 실제 영상 중 생성 영상 영역에 실 제 영상이 많이 존재할수록 Recall이 높다.
이 때, Φr, Φg 는 각각 실제 영상과 생성 영상들의 특징들의 집합이며, ϕr, ϕg 는 집합 Φr, Φg의 특징 원소들이다. NNk는 집합 Φ에 속한 특징 벡터 중 함수의 입력으로 주어 진 특징 벡터 ϕ′ 와 K번째로 가까운 특징벡터를 출력하는 함수이다.
3. 실험 및 결과분석
실험에 사용한 의료 영상 데이터셋은 NSCLC(Non-Small Cell Lung Cancer) Radiomics CT 공 개데이터[24]를 사용하였다. 비소세포폐암 중 선암종 (adenocarcinoma), 편평세포암종(squamous cell carcinoma) 종류의 데이터 190개로 구성되어 있으며, 총 12160장의 슬라이스 영상으로 이루어져 있다. 512 × 512 크기의 흉부 CT 영상을 전체 흉부 영상에서 종양을 중 심으로 160 × 160 크기로 자른 후, 128 × 128 크기로 리 사이징하여 생성 모델의 학습 영상으로 사용하였다.
학습 시 사용한 생성 모델인 DCGAN, PGGAN은 모두 128 × 128 크기의 영상을 생성하며 DCGAN은 64 × 64 크 기의 영상을 생성하는 모델이기 때문에, 128 × 128 크기 의 영상을 생성하기 위해 기존 모델에서 합성곱층을 한 층 추가하였다. 또한, 학습 중 불안정성을 줄이고 최적 화를 위해 생성자의 합성곱층 특징맵 개수가 판별자의 특징맵 개수보다 크도록 조절 후 학습하였다. 또한 학습 중에 손실함수 값이 수렴한 이후에는 학습을 종료하였 다. PGGAN은 모든 층에서 입력 영상의 배치 사이즈를 16으로 고정하였다. DCGAN 모델은 파이토치 공식 홈페 이지에서 제공하는 소스코드를 사용하였고, PGGAN 모 델은 깃허브에 업로드 되어 있는 오픈 소스코드를 사용 하였다.
본 논문에서는 실제 영상과 생성 영상을 동일한 조건 의 영상 크기와 개수 하에 평가하기 위해, 평가 시 생성 영상과 비교될 실제 영상의 크기도 DCGAN과 PGGAN의 생성 영상의 크기에 맞게 128 × 128로 리사이징 하였다. 생성 영상의 수는 슬라이스 총 영상 개수의 50%인 6080개를 생성하고 화소 범위는 (0, 255)로 정규화 후 저장하여 평가데이터를 준비하였으며, 실제 영상도 생성 영상과 동일하게 랜덤하게 6080개로 나누고 화소 범위 도 (0, 255)로 정규화 후 저장하여 기준데이터를 준비하 였다. 또한, 목표로 하는 이상적인 평가 점수값을 측정하 기 위해 실제 영상 중 기준 데이터 6080개를 제외한 나 머지 6080개도 정규화 후 저장하였다. 이 때, 평가 시 기 준 데이터는 실제 영상, 기준 데이터 이외의 실제 영상들 은 생성 영상으로 가정하고 두 영상간의 유사성을 평가하 였으며, 표 1에서 Real의 점수로 나타내었다. 평가 결과의 일반화를 위해 영상 생성 시 유사난수 생성기의 시드 (seed)를 바꾼 잠재공간에서 6080개씩 생성 후 저장하 는 과정을 세 번 반복하였으며, 본 논문의 평가 결과 점 수들은 세 번 반복하여 생성한 영상들의 평가결과에 대 한 총 평균과 표준편차를 반영하였는데, 이 때 표준편차 는 소수점 넷째자리에서 반올림 하였다.
각 모델의 생성한 영상들을 품질과 다양성 측면에서 정량적으로 비교 및 분석하기 위해 1차원 점수기반 평 가방법은 IS, FID를 사용하였고, 2차원 점수기반 평가방 법은 Precision 및 Recall, 개선된 Precision 및 Recall를 사용하여 평가하였다. 모든 평가 지표에서 특징 임베딩 시 사용되는 모델은 파이토치에서 제공하는 ImageNet으 로 사전 학습된 InceptionV3[25]를 파인 튜닝 (fine-tuning)한 모델을 사용하였다. 파인 튜닝 시 튜닝 데이터는 생성 모델 학습 시 사용했던 전체 학습 데이 터를 사용하였으며, 전체 학습 데이터의 클래스는 종양 이 있는 환자의 생존과 사망 두 개로 구성하였다. 모델 보조분류기(Auxiliary classifier)와 마지막 완전연결층 (fully-connected layer)의 출력 클래스 개수는 1000에서 2로 바꾸고, 10 에폭(epoch) 동안 모델의 전체 레이어 중 마지막 출력층을 제외하고 파인튜닝 하였다. InceptionV3 모델은 ImageNet으로 사전 학습 시 299 × 299 크기의 입력 영상이 (-1, 1)로 정규화된 3 채널 의 영상으로 학습되었기 때문에 평가 및 파인 튜닝 시 에도 InceptionV3 모델의 입력 화소 범위는 (-1, 1)의 범 위로 정규화하고, 1채널의 영상을 3채널로 변형하였다. 영상의 크기는 299 × 299로 지정하였는데, 299 × 299 보 다 작은 크기의 영상은 선형 보간(bilinear interpolation) 으로 업샘플링 후 모델의 입력으로 사용하였다. 평가 때 FID, Precision 및 Recall 계열의 경우 InceptionV3 모델 의 2048차원 평균 풀링층에서 특징벡터를 임베딩 하였 다. 실험의 Precision 및 Recall에서 K-평균 군집화를 위 해 사용되는 K값과 조화평균의 가중치인 β값은 Precision 및 Recall[14]에서 실험적으로 사용한 20과 8 을 사용하였으며, 개선된 Precision 및 Recall에서 K-최 근접 이웃 알고리즘을 위해 사용되는 K값은 3을 사용하 였다.
그림 3는 DCGAN과 PGGAN으로 생성한 128 × 128 크기 의 생성 영상 예시로, 육안으로는 어떤 모델이 영상을 더 욱 정확하고 다양하게 생성하였는지 비교하기 어렵다. 그림 4은 그림 3의 생성 영상들과 실제 영상들의 특징을 파 인 튜닝한 InceptionV3 모델에서 임베딩한 뒤 t-SNE 알고 리즘을 통하여 시각화한 결과이다.
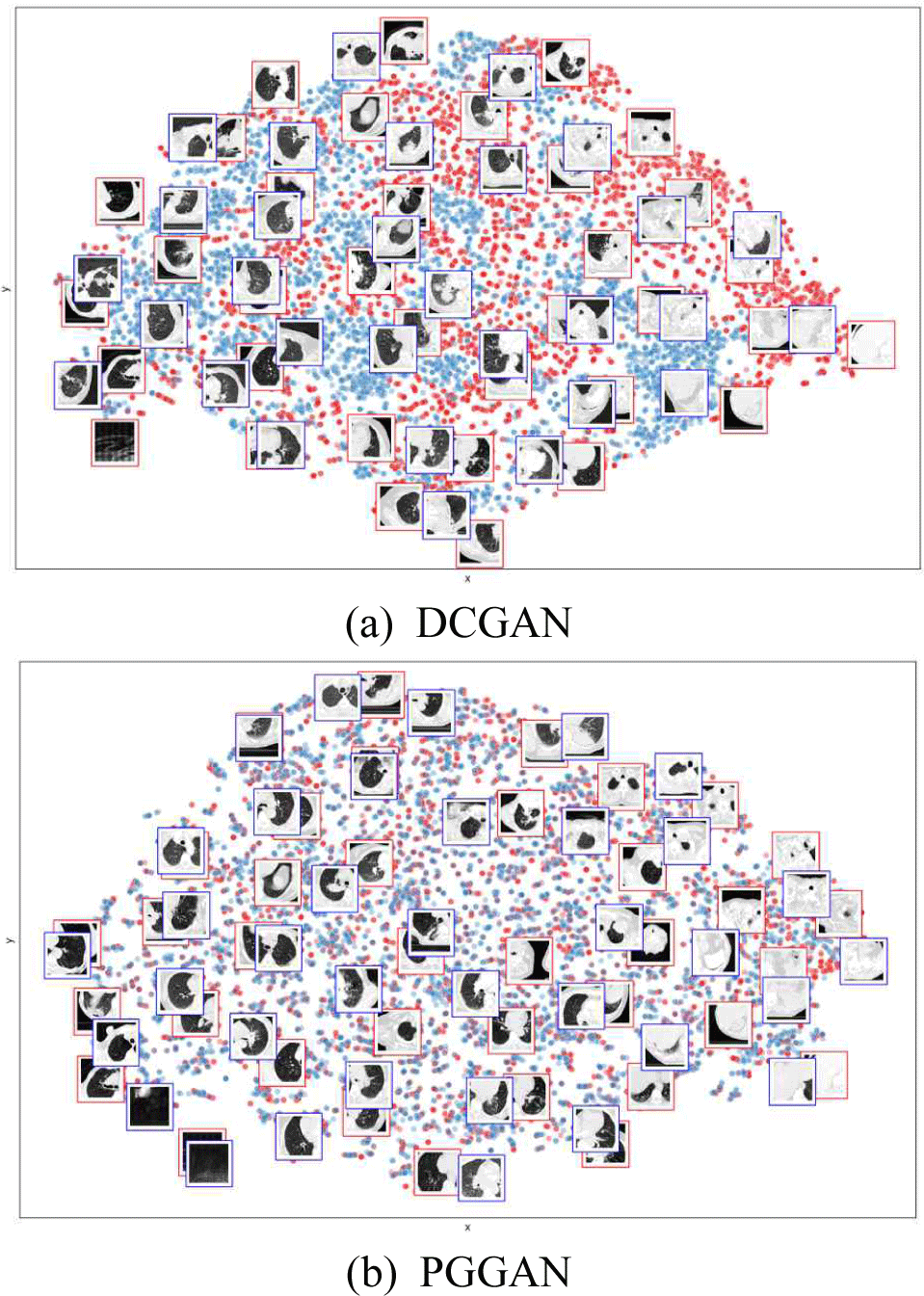
그림 4에서 빨간색 점은 실제 영상들을 나타내며, 파란 색 점은 생성 영상을 나타낸다. 생성 영상의 특징들이 실 제 영상의 분포를 잘 근사하고 있는지 시각적으로 비교하 기 위해, 각 점이 의미하는 실제 영상을 t-SNE의 점 위에 표시하였는데, 이 때, 박스의 테두리가 빨간색인 영상은 실 제 영상, 파란색인 영상은 생성 영상이다. 파인 튜닝 모델 에서의 특징 임베딩 시, t-SNE를 통해 유사한 영상들끼리 가깝게 임베딩 된 것을 알 수 있으며, PGGAN이 DCGAN보 다 실제 영상의 분포를 반영하여 더욱 다양하게 생성이 된 것을 확인할 수 있다.
표 1은 DCGAN과 PGGAN으로부터 생성한 생성 영상들 을 정량적으로 평가한 결과 점수에 대한 평균과 표준편차 이다. 첫째, 1차원 점수기반 평가방법인 IS와 FID를 비교 했을 때, IS의 경우 극소한 차이를 보이고 있으나 FID에서 는 두 모델간의 차이가 IS보다 크게 나타나는 것을 확인할 수 있다. 이는 DCGAN의 생성 영상 분포의 평균은 실제 영상과 유사할지라도 PGGAN보다 DCGAN에서 두 영상 분포 간의 퍼짐 정도의 차이가 크고 다르게 생성이 되었다 는 것을 의미한다. 결국 두 분포 간 표준편차의 차이가 커 지면서 FID 값이 커졌고 DCGAN이 PGGAN보다 실제 영 상 분포를 덜 학습하였다는 것을 나타낸다. 그러나, FID만 으로는 품질과 다양성 측면에서 어떠한 모델이 더욱 정량 적으로 우수한지 분간하기 어렵다. 둘째, 2차원 점수기반 평가방법인 Precision 및 Recall, 개선된 Precision 및 Recall을 비교했을 때, PGGAN이 DCGAN에 비해 품질과 다양성 측면에서 모두 좋게 생성되었음을 알 수 있다. 셋 째, 두 생성 모델의 Precision 및 Recall의 점수는 K-평균 군집화 시 매 계산마다 초기화를 거치기 때문에 항상 평가 결과값이 바뀐다는 한계가 있으나, 개선된 Precision 및 Recall은 K-최근접 이웃을 통해 평가를 반복 했을 때 일 관된 결과값이 유지됨을 알 수 있다. 기존 Precision 및 Recall에서의 K-평균 군집화 방식은 실제 영상과 생성 영 상을 한 번에 묶어서 군집화하기 때문에 상대적인 밀도에 의존성이 발생한다. 이는 두 영상의 영역(manifold) 근사 시 생성 영상의 이상치가 군집화 때 영향을 줄 수 있다는 것을 의미한다. 그러나 개선된 Precision 및 Recall은 두 영상의 영역 근사 시 분포가 아닌 데이터 간의 거리를 구 로 근사함으로써 실제 영상과 생성 영상간의 영향을 주지 않으며, 데이터 각각에 대해 비교하는 방식이기 때문에 모 드붕괴, 다양성이 적은 데이터의 군집과 이상치에 대응할 수 있다.
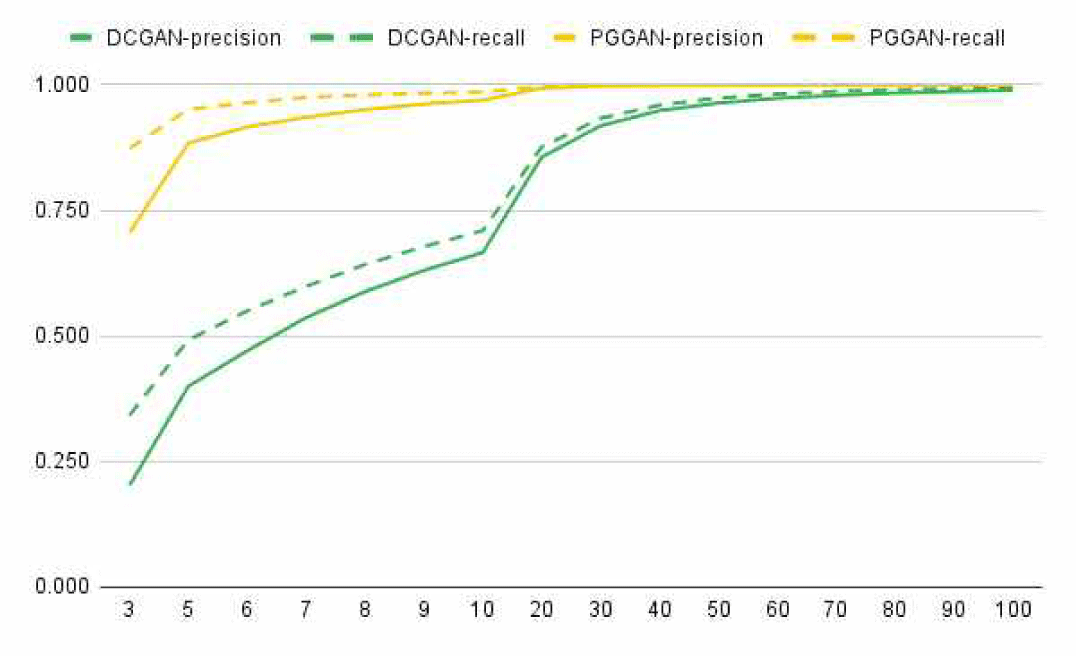
그림 5는 DCGAN과 PGGAN으로부터 생성된 영상들을 K최근접 이웃의 K값에 따라 바뀌는 개선된 Precision 및 Recall 점수를 도표화 하였다. 노란색 선은 DCGAN, 초록색 선은 PGGAN을 의미하며, 실선은 각 모델의 개선된 Precision, 점선은 각 모델의 개선된 Recall을 의미한다. 개 선된 Precision 및 Recall은 특징 공간에서 실제 영상과 생 성 영상들이 임베딩 된 후, K-최근접 이웃 알고리즘에 의 해서 분포의 영역을 근사한다. 이 때 K의 값이 증가할수록 분포의 영역의 크기가 커지면서 실제 영상과 생성 영상의 분포가 겹칠 확률이 증가하게 되고 평가 점수가 증가하게 된다. 그림 5에서는 두 모델 모두 K값이 증가하면 점수가 올라가는 경향을 보이고 있으며, K값이 증가해도 PGGAN 이 DCGAN보다 품질과 다양성 면에서 우수하다는 경향성 을 보이고 있다. 개선된 Precision 및 Recall에서는 실험을 통해 K값을 선정하고 있으므로 데이터 수, 데이터셋 종류 에 따라 이상적인 K값을 선택해야 한다는 것을 알 수 있 다.
4. 결론
본 논문에서는 컴퓨터 비전 분야에서 평가를 위해 사용 되는 1, 2차원 정량적 평가 방법을 통해 생성한 의료 영상 을 품질과 다양성 측면에서 정량적으로 평가하고 결과를 분석하였다. 이를 위해 DCGAN과 PGGAN 모델로 비소세포 폐암이 있는 흉부 CT 영상을 생성하고, 1차원 점수 기반 평가방법인 IS, FID와 2차원 점수 기반 평가방법인 Precision 및 Recall, 개선된 Precision 및 Recall 평가 방법 을 사용하여 평가하였다. IS는 영상의 분포의 차이에 대해 민감하게 변화하지 않는 반면, FID는 영상 분포 차이에 대 해 민감하게 변화하기 때문에 다양성의 차이를 인식할 수 있다. 뿐만 아니라, FID는 모드붕괴 현상 발생을 파악하는 데 용이하지만 다양성의 부재를 품질과 한꺼번에 고려하여 하나의 값으로 출력하기 때문에 세밀한 분석이 어렵다. 이 에 대한 한계점을 2차원 점수 기반 평가방법을 통해 품질 과 다양성을 구분하여 비교할 수 있었다. 그러나 Precision 및 Recall은 K-평균 군집화로 인해 두 영상 분포간의 의존 성이 존재하여 이상치가 군집화 시 영향을 줄 수 있다. 반 면, 개선된 Precision 및 Recall은 분포가 아닌 데이터 각각 을 비교하여 영상의 영역을 근사하고 있기 때문에 의존성 에 영향을 받지 않는다. 그러나 개선된 Precision 및 Recall 은 하이퍼 파라미터인 K값에 의해 이상치가 점수에 영향에 줄 수 있다는 한계점이 있다. 이러한 한계를 개선하기 위 해 최근 Density 및 Coverage[26]가 제안되었고, 추후 연구 에서는 본 연구의 생성 영상을 Density 및 Coverage를 통 해 기존의 평가 방식과 비교 및 분석하고자 한다.
본 연구의 한계점은 다음과 같다. 첫째, 평가를 위한 DCGAN, PGGAN 학습 및 최적화 과정이 쉽지 않다는 점이 있다. GAN은 학습이 불안정하고 손실함수의 수렴이 생성 영상의 좋은 품질 결과를 보장하지 않을 수 있는데, DCGAN의 경우, 단순한 네트워크 구조를 가진 생성 모델 이므로 다른 생성모델들에 비해 낮은 생성 영상의 품질이 평가에 반영된다[16]. 둘째, 컴퓨터 비전에서 IS, FID, Precision 및 Recall, 개선된 Precision 및 Recall은 ImageNet으로 사전 학습된 InceptionV3 모델의 특징공간에 임베딩하여 계산하는데 이 모델의 특징 공간은 ImageNet 에 편향된 공간일 수 있다[16, 27]. 의료영상의 특징을 잘 반영한 특징공간을 구축하려면 대규모의 의료 영상으로 사 전 학습된 모델의 특징공간을 사용하는 것이 적절하나 본 논문에서 사용한 의료 영상 데이터 개수가 적기 때문에 ImageNet에 사전 학습된 모델을 의료 영상에 파인튜닝한 모델의 특징 공간에서 임베딩을 하였다.
본 연구의 향후 연구방향은 다음과 같다. 첫째, 고품질의 영상을 안정적으로 생성할 수 있는 StyleGAN 1, 2[28, 29], BigGAN[30] 등의 다양한 생성 모델로 학습을 진행하고 모 델간의 성능을 비교하고자 한다. 둘째, 대규모의 의료 영상 데이터셋에 사전 학습된 모델을 사용하여 의료 영상 데이 터의 특징을 잘 표현할 수 있는 표현 공간에서 평가 점수 를 비교하고 분석하고자 한다. 셋째, 의료 영상의 표현 공 간에서 고품질로 평가된 의료 영상들을 전문가의 정성적 평가를 통해 검증하는 연구를 진행하고자 한다.