1. 서론
간은 암 전이가 나타나는 대표적인 기관으로[1] 직장암 환자의 15%는 진단 중에 간 전이가 함께 나타나고 50% 이상은 이후 간 전이가 나타나게 되는 것으로 알려져 있다[2]. 진단 또는 치료시기를 놓친 간 전이암의 5년 생존율은 5% 미만으로 알려진 만큼 간 전이암의 조기 발견 및 진단은 암 진단 및 치료에 있어 필수적이다[3]. 암 전이의 조기 발견 및 진단은 주로 컴퓨터단층촬영(Computed tomography; CT) 영상과 같은 의료 영상 분석을 통해 이루어지는데, 전이암(metastasis)의 경우 전문의의 평가를 통해 낭종(cyst)이나 혈관종(hemangioma)과 같은 양성 병변과 감별이 이루어졌다[4]. 최근에는 radiomics와 기계학습 기법을 이용하여 국소 간 병변의 CT 영상으로부터 자동으로 낭종, 혈관종 및 전이암을 분류하려는 시도들이 이루어지고 있다[5].
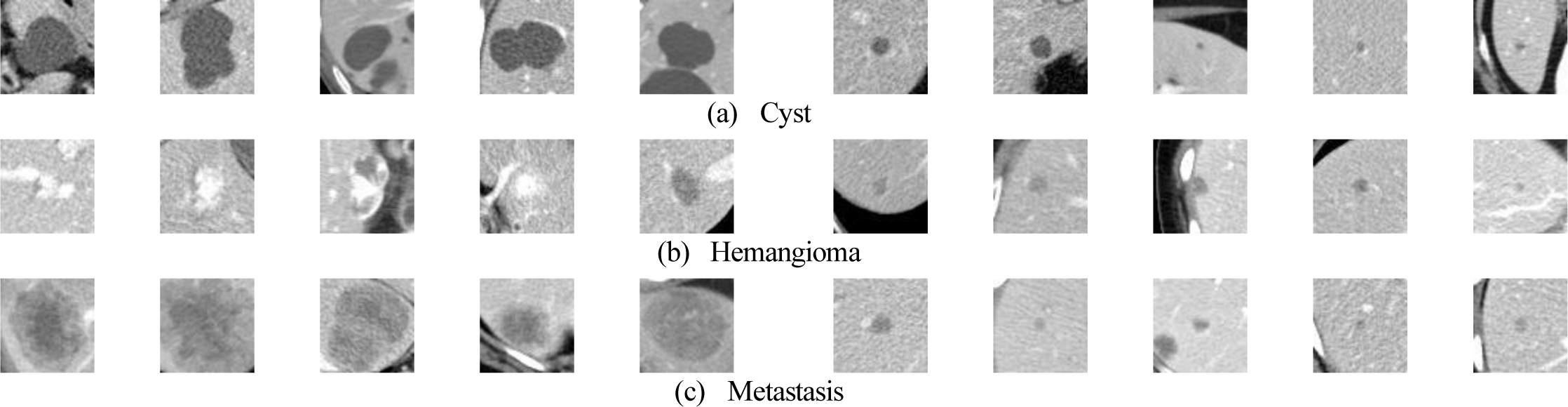
그러나 인공 신경망 등의 학습을 통한 CT 영상에서의 국소 간 병변(Focal Liver Lesion; FLL)의 진단 및 분류에는 몇 가지 어려움이 따른다. 첫째, 데이터가 소량이고, 낭종에 비해 혈관종이 희귀한 클래스 불균형 특성을 갖고 있어 인공 신경망 학습이 어려운 한계가 있다. 둘째, 그림 1과 같이 크기가 작은 병변의 경우 낭종과 혈관종, 전이암의 영상적 특성이 유사하여 신경망 학습 및 분류에 어려움이 있다.
이러한 어려움을 해결하기 위해 데이터 증강(data augmentation) 기법이 사용되는데, 이는 적은 양의 데이터를 바탕으로 다양한 알고리즘을 통해 인위적으로 데이터를 변형함으로써 학습 데이터의 양을 늘려 딥러닝 모델의 과적합(overfitting) 위험을 방지하고, 모델의 일반화(generalization) 성능을 개선하는데 사용된다. 의료 영상에서 데이터 증강 기법은 GAN(Generative Adversarial Network) 학습을 통한 영상 생성과 영상들의 선형 조합을 통해 데이터를 혼합하여 증강하는 혼합 기법 기반의 데이터 증강 기법이 적용되고 있다. GAN 기반 데이터 증강 기법에는 DCGAN(Deep Convolutional Generative Adversarial Network)[6], PGGAN(Progressive Growing GAN)[7], StyleGAN[8] 등이 있다. Salehinejad 등은 흉부 X-ray 영상에서 기흉(pneumothorax), 폐부종(pulmonary edema), 흉막삼출(pleural effusion), 심장 비대(cardiomegaly), 정상(normal)의 5개 영역을 분류하는 문제에서 데이터 증강을 위해 DCGAN 기법을 적용하고, AlexNet을 훈련시켜 분류 성능을 향상시켰다[9]. Frid-Adar 등은 간 병변을 갖는 복부 CT 영상에서 낭종, 혈관종, 전이암의 3가지 질환을 분류하는 문제에서 어파인 변환 기반 데이터 증강 기법을 통해 생성된 영상에 DCGAN을 적용하여 합성곱 신경망(Convolutional Neural Network; CNN)의 성능을 개선하고 분류 성능을 향상시켰다[10]. Zhao 등은 LIDC-IDRI(Lung Image Database Consortium and Image Database Resource Initiative) 흉부 CT 영상에서 폐 결절을 분류하는 문제에서 F&BGAN(Forward and Backward GAN)을 제안하고, VGG16 네트워크의 최대 풀링 계층(max pooling layer) 뒤에 MSB(Multi-Scale Blocks)를 추가한 M-VGG16(Multi-scale VGG16) 네트워크를 훈련시켜 분류 성능을 향상시켰다[11]. Lee 등은 국소 간 병변을 갖는 복부 CT 영상에서 낭종, 혈관종, 전이암의 3가지 질환을 분류하는 문제에서 어파인 변환 기반 데이터 증강 기법과 DCGAN을 사용하여 데이터를 보강하고, pix2pix[12]를 사용한 mask-to-image 변환을 통해 영상과 병변 분할 마스크(lesion segmentation mask)를 모두 생성하여 작은 병변에 대한 CNN의 학습 효율성을 위해 LINA 패치를 제안하고, LINA 패치에 대해 훈련된 CNN 분류기의 앙상블을 통해 분류 성능을 향상시켰다[13]. 이와 같은 GAN 기반 데이터 증강 기법은 기존의 훈련 데이터에 변형을 가하는 어파인 변환 기반 데이터 증강 기법과는 달리 새로운 패턴과 형상의 데이터를 합성하는 강점이 있는 반면, 데이터 증강을 위해 GAN 네트워크를 훈련하는 과정이 필요하고, GAN 네트워크의 생성 효율은 기존 훈련 데이터의 수에 의존하는 것과 특정 패턴의 영상만 생성하게 되는 모드 붕괴 (mode collapse) 현상 등의 한계점이 있다.
혼합 기법 기반 데이터 증강 기법은 영상을 혼합하는 방식에 따라 Mixup[14], Augmix[15], Cutmix[16] 등 다양한 혼합 기법들이 제안되었다. Nishio 등은 흉부 X-ray 영상에서 코로나19 폐렴, 일반 폐렴과 정상으로 다중 분류하는 문제에서 ImageNet 데이터 셋으로 사전 학습된 VGG16 네트워크의 합성곱 계층 (convolution layer)뒤에 전역 평균 풀링(global averaging pooling), 완전 연결 계층과 드롭아웃(dropout)을 적용하였고, 어파인 변환 기반 데이터 증강과 Mixup 기반 데이터 증강을 함께 적용하여 분류 정확도를 높였다[17]. Rajan 등은 흉부 X-ray 영상에서 무기폐(atelectasis), 심장 비대, 폐경화(consolidation), 폐부종, 흉막삼출의 5가지 질환을 분류하는 문제에서 데이터 증강을 위해 어파인 변환 기반 데이터 증강과 Mixup 기반 데이터 증강을 적용하고, ResNet-18 네트워크 훈련 시 일반화 성능 개선을 위해 Mixup Regularization을 적용하여 분류 성능을 향상시켰다[18]. Özdemir 등은 흉부 CT 영상에서 코로나19 확진자와 미확진자를 분류하는 문제에서 Mixup 기반 데이터 증강을 적용하여 기존의 어파인 변환 기반 데이터 증강보다 ResNet50과 ResNet101 모델에서 분류 성능이 개선되는 것을 보였다[19]. 이와 같이 여러 의료 영상 분류 문제에 다양한 데이터 증강 기법을 적용하여 성능 향상을 보인 연구가 이루어졌지만 단일 과제에 여러가지 데이터 증강기법을 적용하여 그 효과를 비교 및 분석하고 검증한 연구는 아직 활발히 수행되고 있지 않다.
본 논문에서는 복부 CT 영상에서의 국소 간 병변 분류를 위한 심층 합성곱 신경망 학습에 다양한 데이터 증강 기법을 적용하여 의료 영상에서의 효과를 평가 및 검증하고자 한다. 이를 위해 어파인 변환 기반 데이터 증강, GAN 기반 데이터 증강 중에서 StyleGAN 기법, 혼합 기법 기반 데이터 증강 중에서 Mixup 및 Augmix 기법을 각각 VGG16 CNN에 적용하여 국소 간 병변을 낭종, 혈관종 및 전이암으로 분류하는 학습을 수행하였다. 각 데이터 증강 기법에 의해 훈련된 모델의 분류 결과에 대하여 분류 정확도(accuracy), 민감도(sensitivity), 특이도(specificity) 수치 분석을 통한 정량적 평가와 증강 영상 예시 분석, tSNE(t-distributed stochastic neighbor embedding)[20] 분석을 통한 특징값 분포도 특성 분석을 통한 정성적 평가를 수행하여 데이터 증강 기법의 효과를 검증 및 분석하였다.
2. 제안 방법
그림 2는 제안한 방법의 개요도를 나타낸다. 먼저, 종양 부분을 중심에 위치시킨 후 크롭(crop)한 64×64 크기의 복부 CT 영상에 어파인 변환 기반 데이터 증강, GAN 기반 데이터 증강 기법인 StyleGAN, 데이터 혼합 기법인 Mixup과 Augmix를 통해 각각 데이터를 증강한다. 둘째, 증강된 데이터를 포함한 훈련 데이터를 ImageNet 데이터셋으로 사전 학습된 변형된 VGG16 모델을 전이학습(transfer learning) 방식으로 학습시킨다. 셋째, 학습된 모델을 사용하여 낭종, 혈관종, 전이암 3개의 클래스로 분류하고, 정확도, 민감도, 특이도 등의 성능 평가를 통한 정량적 평가와 증강 영상 예시 분석 및 tSNE 시각화를 통한 특징값 분포 변화 관찰을 통한 정성적 평가를 수행하여 데이터 증강 기법의 효과를 제시한다.
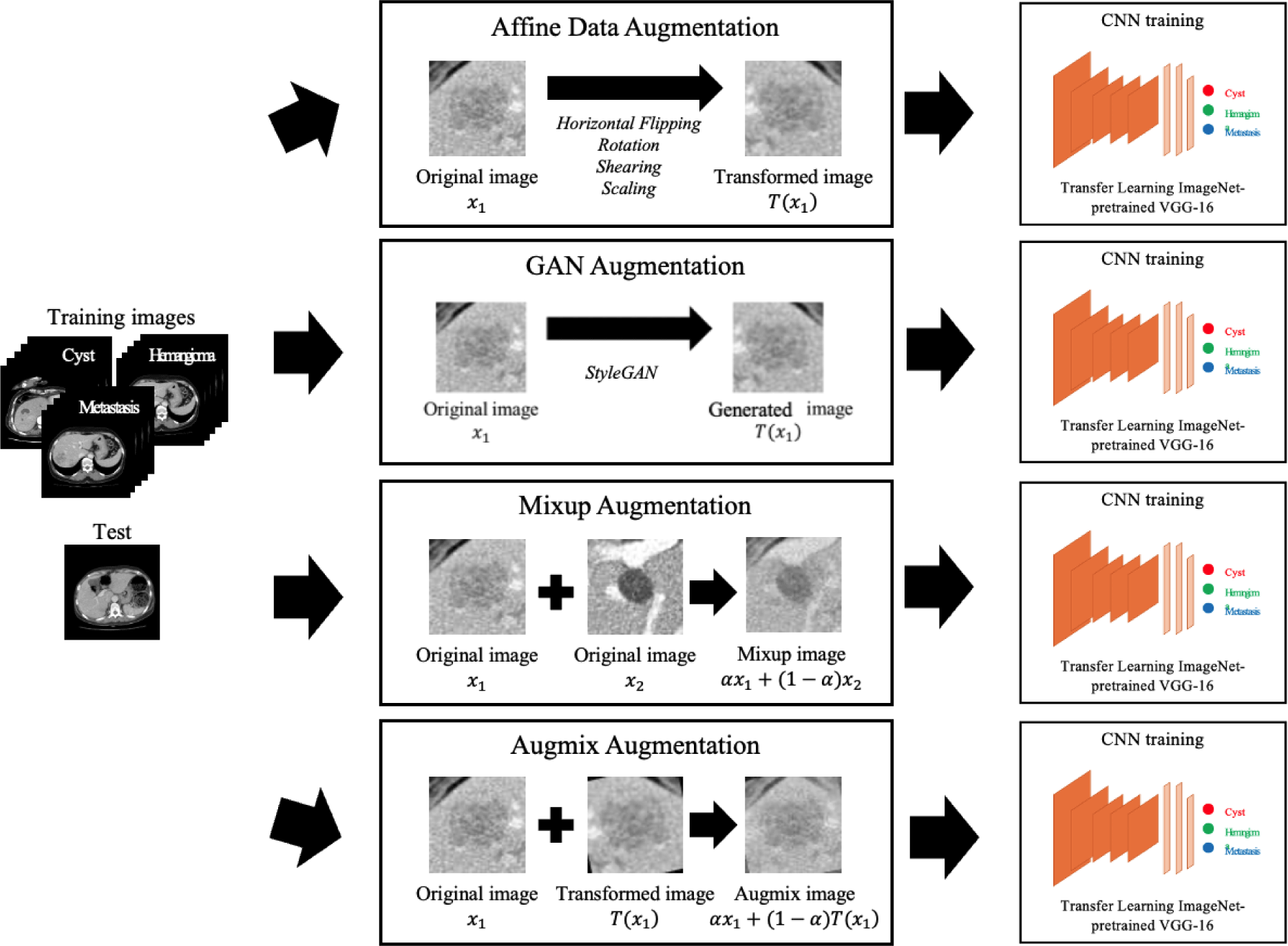
일반적으로 의료 영상 분류 및 분할 문제 등에서 훈련 데이터를 증강시키기 위해 사용하는 어파인 변환 기반 데이터 증강 기법은 이동, 회전(rotation), 수평 뒤집기(horizontal flipping), 자르기, 확대 및 축소 등이 있다[21]. 이와 같은 영상 변환 기반 데이터 증강 기법은 원본 데이터의 공간적 정보를 변형하기 때문에 기하학적 구조가 유지되어 원본 데이터의 속성과 유사하게 데이터가 생성되는 특징이 있다. 따라서, 데이터가 가진 속성들의 다양한 변이를 학습하지 못하여 모델의 일반화 성능 향상에 큰 효과를 보이지 않는 경우가 많다[22].
생성 모델 중 하나인 GAN은 영상을 생성하는 생성기(generator)와 생성한 영상을 판별하는 판별기(discriminator)의 두 개 네트워크를 적대적으로 학습하며 실제 데이터의 분포에 가까운 데이터를 생성한다. 이 때, 생성기는 무작위 잡음(noise)을 초기값으로 입력 받아 영상을 생성하고, 판별기는 입력 영상의 특징들을 학습하여 가짜와 진짜를 0과 1로 판별한 뒤 생성기 네트워크를 업데이트하는 과정을 반복하면, 학습된 생성기에서 임의의 잠재 벡터(latent vector)에 대해 새로운 영상을 만들 수 있게 된다. 의료 영상에서 데이터 증강을 위해 사용하는 DCGAN은 기존의 GAN 구조에 CNN을 적용한 모델로 28×28×3 크기의 상대적으로 작은 크기의 영상을 생성하며, 학습이 불안정하여 모드 붕괴 현상이 발생할 수 있다. 이를 해결하기 위해 손실 함수로 Wasserstein loss를 적용한 후, 4×4의 저해상도부터 1024×1024 고해상도까지 점진적으로 영상을 생성하는 PGGAN은 고해상도 영상을 안정적으로 생성할 수 있다는 장점이 있지만 영상의 특징 제어가 어렵고, 영상의 특징이 분리되어 있지 않기 때문에 다른 특징까지 개입되는 한계가 있다. 따라서 본 절에서는 PGGAN 구조에 style transfer 개념을 적용하여 생성기 구조를 재구성한 StyleGAN을 사용한다.
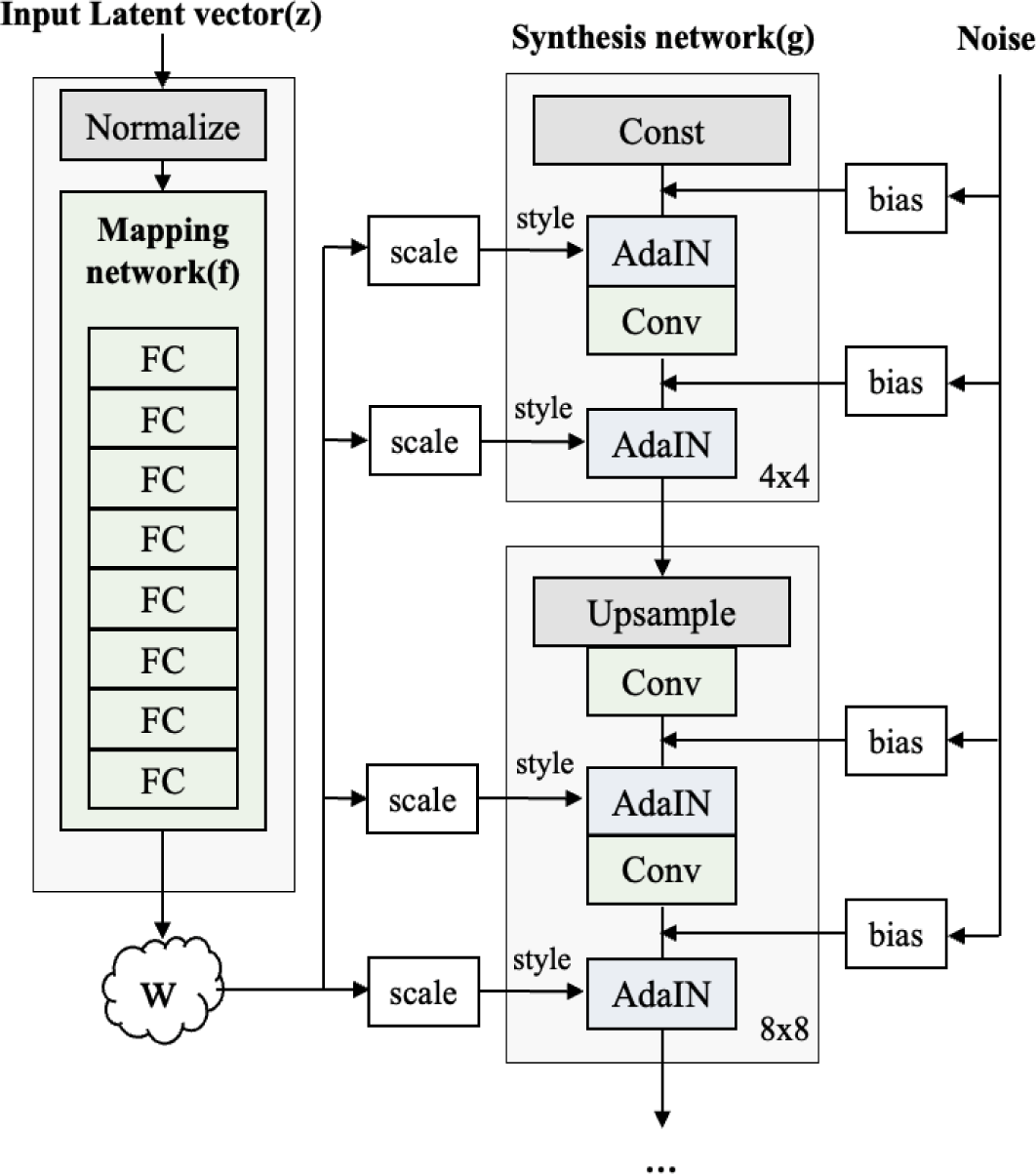
그림 3은 StyleGAN의 네트워크 구조로 입력 벡터 z로부터 직접 영상을 생성하는 것이 아니라 완전 연결 계층 8개로 구성된 매핑 네트워크(mapping network)를 거쳐 중간 잠재 벡터(intermediate latent vector) w로 변환하여 각 스타일들이 잘 분리될 수 있도록 한다. 이 때, w는 합성망(synthesis network)이 영상을 생성하는 과정에서 서로 다른 스타일을 여러 scale에 넣을 때 사용되며, 영상의 다양한 확률적인 측면(stochastic variation)을 바꿀 수 있는 잡음과 함께 어파인 변환 기반 데이터 증강을 거쳐 각 합성곱 계층 이후의 AdaIN(Adaptive Instance Normalization)연산에 사용된다. AdaIN 연산에서 각 층마다 정규화를 수행하고 스타일을 입히는 과정을 통해 최종적으로 64×64 크기의 영상이 생성된다.
이와 같이 GAN 기반 데이터 증강 중 StyleGAN 기법은 매핑 네트워크를 통해 선형성을 가지는 분포가 되어 특징을 분리하기 용이해지고, w는 데이터의 분포에 따라 샘플링 할 필요가 없게 되어 분리된 표현(disentanglement)을 기반으로 보다 현실적인 영상 생성이 가능하다는 장점이 있다.
Mixup[14] 기반 데이터 증강은 수식 1과 같이 서로 다른 클래스에서 무작위로 선정한 두 데이터와 레이블을 선형 보간하여 새로운 데이터를 생성하는 방법이다.
이 때, xi와 xj는 학습 데이터 중 서로 다른 클래스에서 무작위로 선정된 입력 벡터이며 yi와 yj는 각 데이터의 원-핫 레이블 인코딩이다. λ는 베타분포 Beta(α, α)에서 랜덤하게 추출되는 파라미터이며 0에서 1 사이의 값을 가진다. α는 베타분포의 형태를 결정하는 파라미터이다.
Mixup 기반 데이터 증강을 적용하여 클래스의 경계 영역 정보의 강화를 통해 모델의 일반화 오류를 개선하며, 손상된 레이블의 암기를 막고, 적대적 훈련(adversarial training)의 불안정성을 완화시킬 수 있다.
Augmix 기반 데이터 증강은 동일 클래스 내의 영상에 대해 여러 증강 기법들을 직렬 및 병렬로 연결한 후 원본 영상과 다시 혼합하는 방법이다.
그림 4는 Augmix 기반 데이터 증강의 개요도를 나타낸다. 먼저, α=1인 디리클레 분포로부터 합이 1이 되는 k개의 가중치 w1-부터 wk 를 추출하고, k개의 데이터 증강 기법을 랜덤하게 선택하여 그 중 하나 이상의 데이터 증강 기법을 랜덤으로 조합하여 k개의 augmentation 체인을 형성한다. 본 논문에서는 k=3으로 실험을 통해 설정하고, 회전, 수평 뒤집기, 확대 및 축소, 밀림의 증강 기법을 랜덤으로 조합하는데 사용한다. 둘째, 원본 영상인 xorig를 k개의 augmentation 체인을 통해 k개의 영상으로 생성한 후, 대응하는 가중치의 값만큼 혼합하여 xaug를 만든다. 마지막으로, 수식 2와 같이 베타분포 Beta(α, α)로부터 추출된 가중치 m의 비율만큼 xorig와 xaug를 Mixup하여 새로운 영상인 xaugmix를 생성한다.
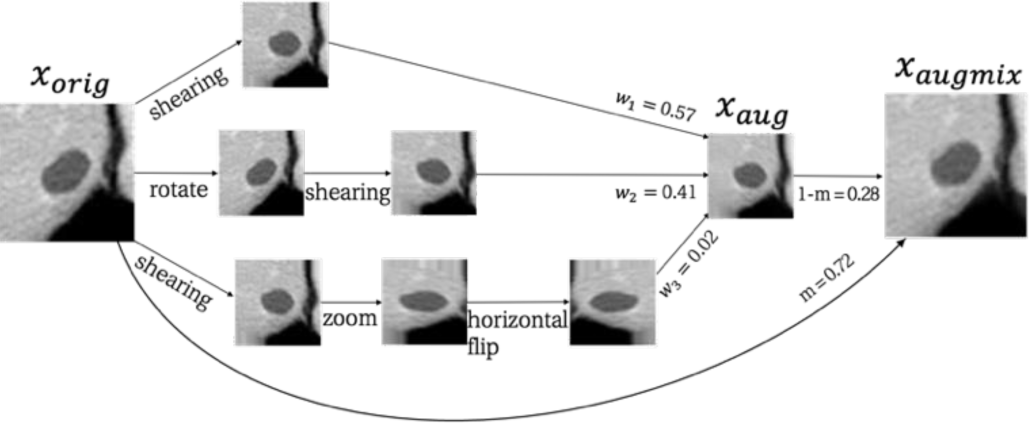
이 때, m은 베타분포 Beta(α, α)에서 랜덤하게 추출되는 파라미터이며 0에서 1 사이의 값을 가진다.
Augmix 기반 데이터 증강을 적용하여 자기 데이터와의 선형 조합을 통해 더욱 안정된 데이터 증강을 할 수 있고, 모델의 강건성(robustness)을 높이고 불확실한 추정을 향상시킬 수 있다.
국소 간 병변 분류를 위해 ImageNet 데이터 셋에서 사전 학습된 분류기인 VGG16 네트워크를 변형[17]하여 학습에 사용한다. VGG16 네트워크는 13개의 합성곱 계층과 3개의 완전 연결 계층으로 이루어져 있으며, 모든 합성곱 계층에서 3×3 필터를 사용한다. 본 연구에서는 VGG16의 합성곱 계층 뒤에 전역 평균 풀링, 완전 연결 계층과 드롭아웃을 연결한다. 이 때, 드롭아웃 계층의 확률은 0.1로 지정하고, 마지막 분류기 직전단의 완전 연결 계층의 노드 수는 기존 4096에서 416으로 변경한다. 낭종, 혈관종, 전이암 3개의 클래스로 분류하기 위하여 드롭아웃 계층 뒤에 3-유닛 완전 연결 계층을 추가하고, 마지막 연결 계층에 소프트맥스(softmax) 함수를 적용한다. 활성화 함수로는 ReLU 함수를 사용하며, VGG16 합성곱 계층 앞 단의 약 55%를 동결하여 전이 학습을 진행한다.
3. 실험 및 결과 분석
본 연구에서는 세브란스병원에서 기관생명윤리위원회 (IRB)의 승인을 받아 2005년 1월부터 2010년 12월 사이에 502명의 대장암 환자로부터 획득한 1290개의 복부 CT 영상[23]을 사용하였고, 데이터 셋의 구성은 표 1과 같다. 모든 데이터는 단일 기관의 다중 검출 CT(multidetector CT; MDCT)를 통해 수집되었으며 사용된 MDCT는 4채널 MDCT (LightSpeed Plus, GE Healthcare, Milwaukee, WI, USA), 16채널 MDCT (Somatom Sensation 16, Siemens Healthcare, Erlangen, Germany; LightSpeed VCT, GE Healthcare, Milwaukee, WI, USA), 그리고 64채널 MDCT (Somatom Sensation 64, Siemens Healthcare, Erlangen, Germany; Somatom Definition Flash, Siemens Healthcare, Erlangen, Germany)이다. 모든 스캔의 절편 두께(slice thickness)는 3~5 mm이고, 해상도는 512×512 픽셀이며, 픽셀 크기는 0.5 × 0.5 mm2~ 0.8 × 0.8 mm2이다. 영상 전처리 단계에서 모든 스캔의 크기가 0.8 × 0.8 mm2 픽셀의 크기로 조정되었다.
| Cyst | Hemangioma | Metastasis | Total | |
|---|---|---|---|---|
| Training Dataset | 433 | 70 | 178 | 681 |
| Validation Dataset | 115 | 30 | 157 | 302 |
| Test Dataset | 128 | 30 | 149 | 307 |
| Total | 676 | 130 | 484 | 1290 |
낭종, 혈관종, 전이암을 갖는 훈련 영상에 어파인 변환 기반 데이터 증강, StyleGAN, Mixup, Augmix 기반 데이터 증강을 각각 적용하고 학습시킨 뒤 분류 결과를 비교했다. Mixup과 StyleGAN 모델의 경우 어파인 변환 기반 데이터 증강을 함께 적용하였다.
어파인 변환 기반 데이터 증강 기법으로는 ± 15° 회전, 수평 뒤집기, 85–115% 확대 및 축소, ± 10% 밀림을 적용하였다. StyleGAN 기반 데이터 증강에서의 하이퍼파라미터는 반복(itetation) 70000, 배치 사이즈(batch size) 8, 학습률(learning rate) 0.001로 설정하였고, 생성 영상은 각 클래스 훈련 영상의 수와 동일하다. StyleGAN 기반 데이터 증강 기법으로 생성된 영상을 평가하기 위해, 원본 영상과 StyleGAN 생성 영상을 미니배치(mini batch) 안에서 일정 비율로 영상의 수를 섞은 후, 어파인 변환 기반 데이터 증강 기법을 사용하여 성능을 평가하였는데, 비율은 원본 영상에 StyleGAN 생성 영상이 전혀 포함되지 않은 0%부터 25%, 50%, 75%, 100%까지의 총 5가지 비율을 각각 적용하였다. Mixup 기반 데이터 증강에서의 α는 0.1~1.0을 적용하여 실험하였고, Augmix의 augmentation 체인 형성에 사용되는 데이터 증강 기법은 어파인 변환 기반 데이터 증강과 동일하게 ± 15° 회전, 수평 뒤집기, 85~115% 확대 및 축소, ± 10% 밀림으로 설정하였으며, Augmix에서의 α는 0.1~0.5를 적용하여 실험하였다.
어파인 변환 기반 데이터 증강과 StyleGAN, Mixup, Augmix 기반 데이터 증강을 적용한 학습 네트워크의 하이퍼파라미터는 에폭(epoch) 100, 배치 사이즈 8, 학습률 0.0002, 훈련 조기 종료(early stopping) 조건을 20으로 설정하였다.
본 논문에서는 다양한 데이터 증강 기법에 따른 간 전이암 분류의 신경망 분류 성능 및 효과를 정량적 및 정성적 평가를 통해 분석하였다. 간 전이암 분류 효과 분석을 위한 정량적 성능 평가 방법으로는 각 모델에 대한 정확도와 낭종, 혈관종, 전이암 각각에 대한 민감도, 특이도를 사용하였다. 모델의 강건성을 보장하기 위하여 어파인 변환 기반 데이터 증강, StyleGAN, Mixup, Augmix 각 모델별로 정확도, 민감도, 특이도의 평가 결과를 5개의 모델에서 계산하여 그 평균과 표준편차를 구하였다. 정성적 평가 방법으로 증강 영상 예시 분석과 데이터의 고차원 특징을 차원 축소하여 2차원으로 시각화하는 tSNE를 사용하였고, VGG16 네트워크의 마지막 완전 연결 계층에서 나온 데이터의 특징을 tSNE 알고리즘을 통해 시각화 함으로써 각 데이터 증강 기법에 따라 훈련된 분류기를 통해 추출된 훈련 데이터, 증강 훈련 데이터 및 시험 데이터의 특징값 분포를 관찰함으로써 분류 효과와 성능을 분석하였다.
그림 5는 낭종, 혈관종, 전이암 영상 중 원본 영상, StyleGAN 기반 데이터 증강을 통해 생성한 영상, Mixup 및 Augmix 기반 데이터 증강을 통해 생성한 영상을 나타낸다. 그림 5의 (d), (e), (f)는 StyleGAN 기반 데이터 증강을 통해 생성된 영상이다. 어파인 변환이나 Mixup, Augmix 기반 데이터 증강 기법은 기존 영상을 변형하는데 그치는데 반해 StyleGAN으로부터 생성된 영상들은 새로운 조합이 가능하다. 하지만 영상을 생성하는데 있어서 한정된 패턴이 계속해서 나오는 모드 붕괴 현상이 발생한다는 한계가 있다. 그림 5의 (g), (h), (i)는 Mixup 기반 데이터 증강을 통해 생성된 영상이다. 비교를 위해, 12개의 낭종, 혈관종, 전이암 영상에 모두 동일한 혈관종 영상을 혼합하였다. λ 값이 1에 가까울 수록 원본 영상의 특성을 많이 가지고 있으며, 0에 가까울 수록 혼합된 혈관종 영상의 특성이 드러나는 것을 볼 수 있다. 그림 5의 (j), (k), (l)은 Augmix 기반 데이터 증강을 통해 생성된 영상이다. Augmix는 마지막 연산에서 원본 영상과의 혼합이 이루어지기 때문에 생성 영상이 원본 영상과 거의 유사한 것을 볼 수 있다.

표 2는 어파인 변환 기반 데이터 증강, StyleGAN, Mixup 그리고 Augmix 기반 데이터 증강을 사용하여 증강된 데이터로 학습된 각 모델의 평가 결과에 대한 평균과 표준편차이다. StyleGAN 모델에서는 StyleGAN으로부터 생성한 영상들을 원본 영상과 일정한 비율로 섞어서 성능을 평가했으며, 원본 영상에 StyleGAN으로부터 생성된 영상을 0%, 25%, 50%, 75%, 100%의 비율로 섞었을 때 25%의 비율에서 가장 높은 분류 정확도를 보여 해당 모델을 평가에 사용하였다. Mixup 모델에서는 α가 0.1~1.0 중 0.6인 경우에 가장 높은 정확도를 보여 α = 0.6인 경우의 모델을 평가에 사용하였고, Augmix 모델에서는 α가 0.1~0.5 중 0.1인 경우에 가장 높은 정확도를 보여 α = 0.1인 경우의 모델을 평가에 사용하였다.
StyleGAN 모델의 경우, 평균 정확도가 73.42%로 어파인 변환 기반 데이터 증강에 비해 0.85% 높게 나타났다. 혈관종의 민감도와 특이도, 전이암의 민감도와 특이도 또한 어파인 변환 기반 데이터 증강에 비해 각각 6.0%, 1.45%, 0.54%, 0.76% 높은 수치의 결과를 보였다. 적은 수의 원본 데이터를 단독으로 사용하는 것 보다는 StyleGAN으로부터 생성된 영상을 25%의 비율로 함께 사용하는 것이 전반적인 성능을 보완하는 효과가 있었다. Mixup 모델의 경우, 평균 정확도가 74.13%로 어파인 변환 기반 데이터 증강에 비해 1.56% 높게 나타났다. 낭종의 민감도와 혈관종의 특이도, 전이암의 민감도 또한 어파인 변환 기반 데이터 증강에 비해 각각 2.03%, 5.92%, 2.28% 높은 수치의 결과를 보였다. Augmix 모델의 경우 정확도, 민감도, 특이도가 어파인 변환 기반 데이터 증강에서 보다 전체적으로 떨어지는 경향이 나타났으나 혈관종의 민감도와 전이암의 특이도의 경우 어파인 변환 기반 데이터 증강에 비해 각각 10%, 1.23% 높은 수치의 결과를 보였다.
그림 6은 각 데이터 증강 기법에 따라 훈련된 분류기를 통해 추출된 훈련 데이터, 증강 훈련 데이터 및 시험 데이터의 특징값의 분포를 tSNE 기법을 통해 시각화한 것이다. 그림 6 (a)와 (b)에서 보듯이 주어진 데이터는 낭종과 전이암이 양 끝에 분포하고 그 경계 영역에 혈관종이 겹쳐서 나타나는 분포를 보인다. 이러한 분포는 낭종의 분류 성능은 높은 반면 혈관종과 전이암의 분류 성능은 상대적으로 떨어지고, 혈관종과 전이암 중에서도 데이터의 개수가 적은 혈관종의 분류 성능이 상대적으로 더 떨어지는 현상을 설명할 수 있다.
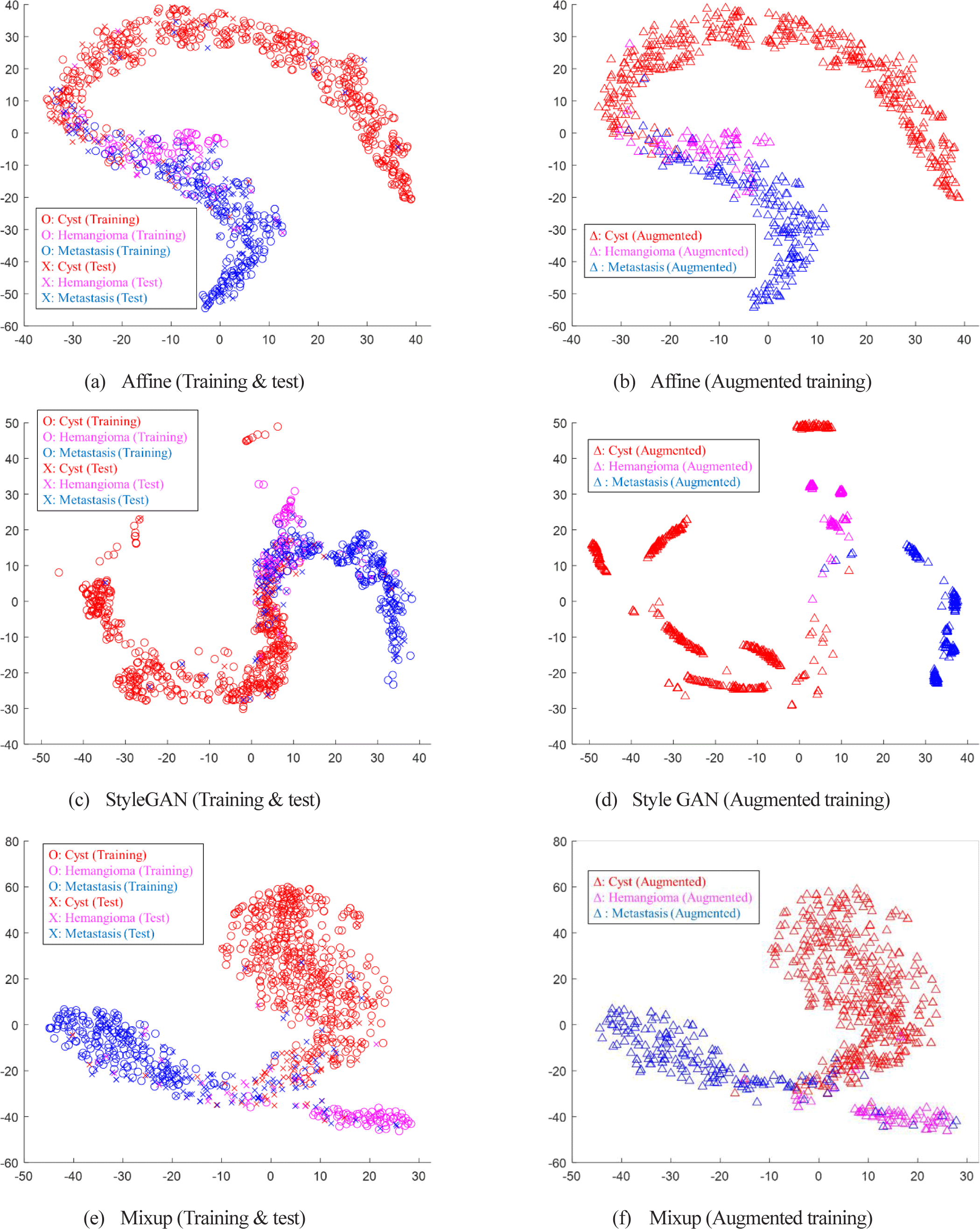
그림 6 (c)와 (d)는 StyleGAN 기반 증강 25% 포함 데이터에 의해 훈련된 분류기에서 추출된 훈련 및 시험 데이터와 StyleGAN 기반 증강 데이터의 tSNE 분포를 나타낸 것이다. 앞의 어파인 변환 기반 증강 데이터가 훈련 데이터의 전체 분포를 복제하는 형태의 증강 특성을 보인 반면 StyleGAN 기반 증강 데이터는 각 클래스에 대해 경계 영역의 반대편 극단의 데이터에 집중적으로 분포하는 특성을 보인다. 이는 GAN이 클래스의 특성이 혼재되거나 모호한 경계 영역의 영상이 아니라 각 클래스의 특성이 분명하게 나타나는, 분류가 용이한 가상 영상을 생성하도록 학습되기 때문에 이와 같이 다른 클래스들과 가장 먼 영역의 영상들이 생성되는 것으로 분석될 수 있다.
그림 6 (e)와 (f)는 Mixup 기반 증강 데이터에 의해 훈련된 분류기에서 추출된 훈련 및 시험 데이터와 증강 훈련 데이터의 tSNE 분포를 나타낸 것이다. 경계 영역의 반대편을 강화한 StyleGAN과는 달리 Mixup 기반 증강 데이터는 실제 훈련 데이터에 비해서 세 클래스의 경계 영역에 집중적으로 분포하는 특성을 보인다. 이는 Mixup이 서로 다른 클래스 영상들의 선형 조합을 통해 두 클래스의 중간 영역에 해당하는 데이터들을 생성하고 연성라벨 생성과 일관성 정규화를 통해 이러한 경계 영역 정보를 강화한다는 설명과 일치한다. 이러한 경계 영역 정보의 강화를 통해 Mixup은 주어진 과제에서 가장 높은 분류 정확도를 달성하였다.
그림 7은 Augmix 기반 증강 데이터에 의해 훈련된 분류기에서 Augmix 기반 증강 데이터와 어파인 변환 기반 증강 데이터의 tSNE 분포를 비교한 것이다. Augmix의 증강 특성은 기존 훈련 데이터 분포 전체를 복제하는 것으로 보인다는 점에서 앞의 어파인 변환 기반 데이터 증강의 특성과 거의 동일한 특성을 보인다. 그러나 어파인 변환 기반 증강 데이터의 경우 낭종 영역 안에 있는 전이암 데이터와 같이 클래스 영역을 벗어난 증강 데이터가 Augmix 에 다수 발견된다. 이는 데이터에 심한 어파인 변환 기반 데이터 증강을 가해서 클래스 영역을 벗어날 수도 있는 데이터들에 대해서 Augmix는 자기 데이터와의 선형 조합을 통해 더욱 안정된 데이터 증강 특성을 보이는 것으로 분석할 수 있다.
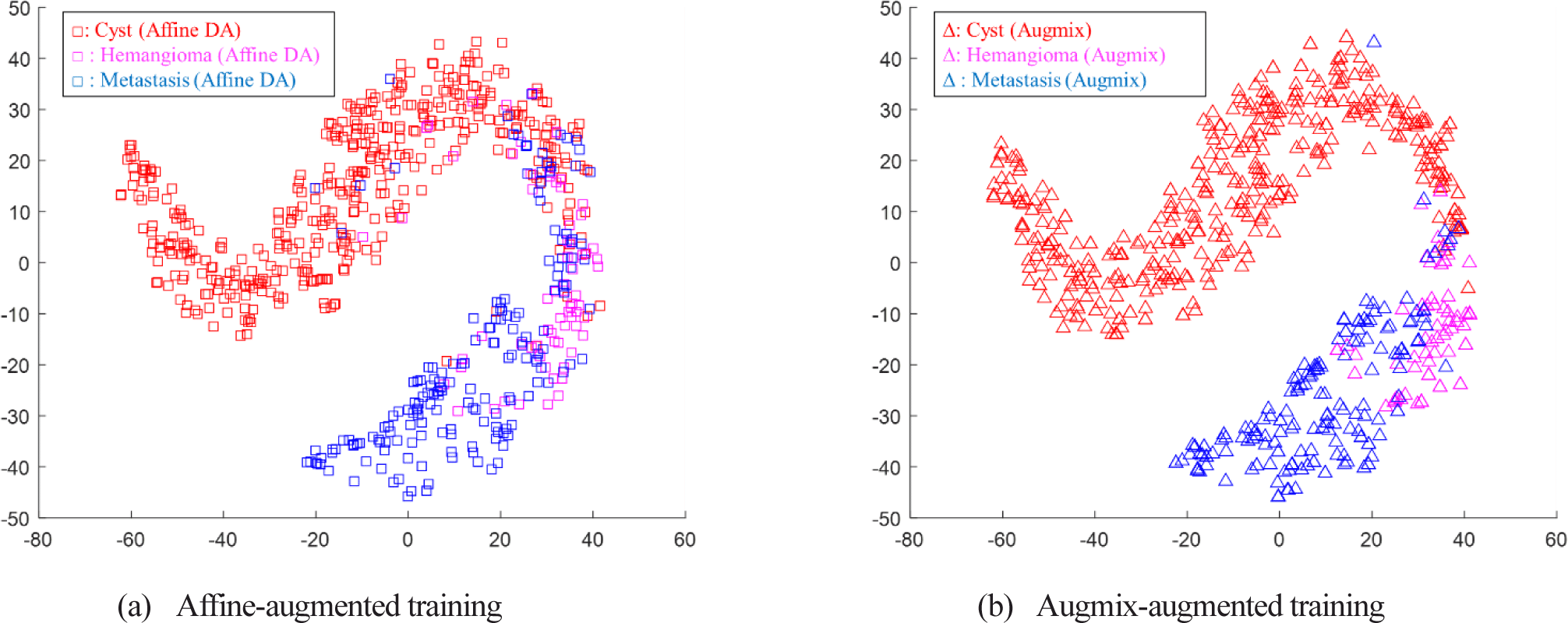
따라서, 다음과 같이 각 데이터 증강 방법의 특성을 정리할 수 있다. StyleGAN 생성 데이터는 경계 영역의 영상 생성 보다는 각 클래스의 특성이 분명하게 나타나 새로운 조합의 영상이 필요한 영상을 증강할 때 적합하다. Mixup의 경우 세 클래스 사이의 경계 영역에 집중적으로 분포함으로써 증강 기법 중에 가장 큰 정확도 개선을 보여 안정적으로 다중 클래스의 영상을 증강할 때 적합하다. Augmix는 안정된 데이터 증강 특성을 보임으로써 클래스 내부를 강화하는 특성을 보여 소수 클래스의 영상을 증강할 때 적합하다. 본 연구에서 사용된 국소 간 병변 데이터는 데이터가 소량이며 낭종에 비해 혈관종이 희귀한 클래스 불균형 특성을 가진다. 이러한 데이터 증강 방법과 데이터의 특성을 바탕으로, 국소 간 병변 데이터의 다중 클래스 분류 문제에서는 Mixup 기반 증강 방법이 가장 좋은 효율을 보이는 것으로 나타난다.
4. 결론
본 연구에서는 복부 CT 영상에서 국소 간 병변 분류를 위한 심층 합성곱 신경망 학습에 다양한 데이터 증강 기법을 적용하여 의료 영상에서의 효과를 평가 및 분석하였다. 이를 위해 어파인 변환 기반 데이터 증강, GAN 기반 데이터 증강 중에서 StyleGAN 기법, 혼합 기법 기반 데이터 증강 중에서 Mixup 및 Augmix 기법을 각각 VGG16 CNN에 적용하여 국소 간 병변을 낭종, 혈관종 및 전이암으로 분류하는 학습을 수행하였다. 정량적 평가에서는 각 데이터 증강 기법에 의해 훈련된 모델의 분류 결과에 대하여 분류 정확도, 민감도, 특이도 수치 분석을 수행하였으며, 정성적 분석에서는 증강 영상 예시 분석, tSNE 분석을 통한 특징값 분포도 특성 관찰을 통해 데이터 증강 기법의 효과를 분석하였다. 정량적 평가에서 StyleGAN은 전반적으로 어파인 변환 증강 기법과 유사한 결과를 보였으며, Mixup은 낭종과 전이암의 민감도에서, Augmix는 혈관종의 민감도에서 개선된 결과를 보였다. tSNE 특징값 분포도 관찰을 통한 정성적 분석에서 StyleGAN 생성 데이터는 각 질병 클래스의 특징이 뚜렷한 외곽 영역에 주로 분포함으로써 세 클래스의 분류 성능을 결정하는 경계 영역의 개선에 큰 역할을 하지 않으며, Mixup의 경우 세 클래스 사이의 경계 영역을 집중적으로 강화함으로써 증강 기법 중에 가장 큰 정확도 개선을 보인 반면 소수 클래스인 혈관종의 민감도가 떨어지는 한계를 보였고, Augmix는 어파인 변환과 유사하면서도 안정된 데이터 증강 특성을 보임으로써 클래스 내부를 강화하는 특성을 보여 소수 클래스인 혈관종의 민감도를 개선하는 결과를 보였다. 이와 같은 증강 기법의 효과 및 증강 특성 분석을 통하여 주어진 문제와 데이터셋의 특성에 맞는 증강 기법을 선택적으로 적용하거나, 서로 다른 특성을 갖는 증강 기법을 함께 적용함으로써 그 효과를 보완하게 하는 데 활용할 수 있다.