1. 서론
최근 실시간으로 복잡한 삼차원 시각화를 구현하려는 많은 연구들이 활발히 진행되고 있다. 특히, 신경망 기반의 영상 생성 모델과 물리 시뮬레이션 기술의 결합은 고해상도 모델의 현실적인 시각화를 가능하게 한다. 이는 신경망과 물리 시뮬레이션 유한 요소법을 사용하여 실제 환경에서 실시간으로 뇌 종양의 위치를 추적하거나, 양악 수술 후 안면의 변형을 예측하여 시각화하는 등 의료 분야에서 많은 연구들이 존재한다[1, 2].
딥러닝(Deep Learning)은 의료 분야에서 영상 진단, 생체 의학, 임상 데이터 분석 및 분류 등 다양한 방면에서 사용된다[3-5]. 딥러닝 기법의 일종인 신경 방사 필드(Neural Radiance Fields, NeRF)는 이차원 영상집합과 각 영상에 대한 카메라의 자세와 방향을 입력으로 학습을 수행하여 새로운 시점에서 장면을 바라본 영상을 생성한다[6-8]. 입력으로 사용되는 영상집합은 핸드폰 카메라와 같이 쉽게 이용할 수 있는 기기들을 시작으로 하여, CT, MRI 또는 CBCT등 고가의 삼차원 의료영상 장비까지 다양한 형태로 획득하여 사용할 수 있다.
인체 내부 장기를 다루는 수술 계획은 높은 정밀성을 요구한다. 이는 일반적으로 CT나 MRI등의 삼차원 의료영상 장비로부터 환자의 시술 부위에 대한 데이터를 획득한다. 그러나 이러한 장비는 방사선 피폭과 긴 촬영 시간 등의 단점이 있다[9]. 최근 NeRF는 CT, MRI 또는 CBCT등의 삼차원 의료영상 장비로부터 환자의 시술 부위를 획득한 데이터의 일부만을 영상집합으로 사용하여 동일한 볼륨 데이터를 생성하는 연구들이 진행되고 있다[10, 11]. 그리하여 긴 촬영 시간을 단축시키고 그에 따른 방사선 피폭을 최소화한다.
한편, 본 연구에서 볼륨 데이터의 변형에 사용하는 위치 기반 동역학(Position-Based Dynamics, PBD)는 속도와 정밀성을 모두 고려한 균형 잡힌 물리 기반 시뮬레이션 기법이다. 외력에 의해 변형된 물체의 움직임을 제약 조건에 따라 정점에 대한 직접적인 수정을 가하여 움직임을 제어하는 특성을 기반으로, 안정적인 실시간 변형과 상호작용을 가능하게 한다[12]. 이는 가상 의료 시뮬레이션 프로그램이 가져야 할 빠른 속도와, 실제와 같은 대상의 움직임을 표현하는 요구를 충족시킨다. 또한, PBD는 다른 물리엔진들에 비해 구현이 간단하고 효율적이다. 게다가 PBD는 병렬 처리를 효과적으로 활용할 수 있는 구조를 가지고 있다[13].
의료영상과 같은 고해상도 데이터에 대하여, 사용자가 상호작용을 통해 실시간으로 변형을 수행하고, 변형된 결과를 가시화하기 위한 빠른 연산속도는 필수적이다. Aguilera가 제안한 병렬 리샘플링은 효율적인 GPU 병렬화를 통해 변형 연산과 그 결과로 생성된 변형 볼륨 데이터 가시화를 가속화한다[14]. GPU 병렬화를 이용하는 다양한 연구들[15, 16]이 존재하나 병렬 리샘플링은 묵시적 샘플링 메쉬를 적용하여 변형 연산을 래스터화(rasterization) 과정에서 분리하고, 내부적으로 사용되는 변형 구조의 해상도에 의존하지 않는다. 이는 다양한 해상도의 데이터에서 동일한 변형 연산을 적용할 수 있도록 한다.
본 연구에서는 NeRF로 삼차원 볼륨 데이터를 효과적으로 생성하고 PBD를 이용하여 변형하며 병렬 리샘플링을 이용하여 변형된 데이터를 생성하고 가시화하는 결합된 시스템을 제안한다. 최근의 딥러닝 연구는 데이터 변형 과정에서도 학습을 통하여 데이터를 생성하려는 반면, 본 연구는 일반적인 물리 기반의 변형과 변형체 가시화 기법을 적용하여 하나의 프레임워크에서 볼륨 데이터의 생성과, 변형, 가시화를 GPU 기반의 CUDA[17, 18]로 구현하였다. 이와 같은 구성은 NeRF, PBD 그리고 병렬 리샘플링의 결합을 통한 고해상도 삼차원 시각화 기술의 가능성을 탐구하고, 이를 통해 향후 의료영상 및 다양한 응용 분야에서의 실질적 적용 가능성을 제시한다.
본 연구의 구성은 다음과 같다. 2장에서는 본 연구에서 사용하는 딥러닝 모델과 변형체 가시화 관련 연구를 설명한다. 특히, 이 장에서는 딥러닝과 물리 기반 시뮬레이션 기술의 결합에 초점을 맞추어 설명하고 이와 관련된 이전 연구를 살펴본다. 3장에서는 제안 시스템의 구현 방법에 대해 설명하고, 4장에서는 NeRF 모델을 기반으로 한 삼차원 볼륨 데이터 생성과 PBD를 이용한 실시간 변형체 가시화에 대해 다룬다. 5장에서는 실험 결과를 보이고, 6장에서 결론을 맺는다.
2. 관련 연구
NeRF는 공간상의 각 위치(x, y, z)에서 특정 방향(θ, φ)으로 방출되는 광도(radiance)와 밀도(density)를 나타내는 연속적인 5차원 공간에서의 함수로 장면을 표현한다[6]. 여기서 밀도는 광선이 해당 위치를 통과하면서 누적된 광도(radiance)의 양을 표현하는 불투명도를 나타낸다. NeRF는 완전연결 신경망의 일종인 다층 퍼셉트론(Multi-Layer Perceptron, MLP)으로 학습을 수행한다. 이를 이용하여 NeRF는 5차원 좌표(x, y, z, θ, φ)에 대한 밀도와 관측 방향에 따른 RGB색상을 추정한다.
관측 방향에 따른 색상 값을 추정하기 위해, NeRF는 이차원 영상집합의 각 영상에 대한 카메라 포즈 정보를 필요로 한다. NeRF는 이 정보를 삼차원 장면을 재구성하는 Structure-from-Motion(SfM) 기술을 사용하여 획득할 수 있다[19]. SfM은 다양한 시점에서 촬영된 영상들을 이용하여 장면의 삼차원 공간을 추정하고, 그 과정에서 카메라의 자세와 방향 정보를 계산한다.
PBD 알고리즘은 물리 기반 시뮬레이션에서 널리 사용되는 기법으로 Müller는 PBD의 기본 원리를 제시하였다[12]. PBD는 물체의 위치를 수정하여 제약 조건을 만족시키는 방식으로, 물리적 정확성을 유지하면서 계산 효율성을 높일 수 있다. Macklin은 PBD를 확장하여 더 복잡한 물리적 현상을 모델링할 수 있는 기법을 제안하였다[13, 20]. 이는 PBD를 이용하여 유체, 고체, 섬유 등의 다양한 물질을 실시간으로 시뮬레이션할 수 있음을 보인다. 또한, PBD의 확장된 형태인 XPBD(Extended Position-Based Dynamics)를 도입하여, 기존 PBD의 한계를 극복하고 더 정밀한 시뮬레이션을 가능하게 하였다. Bender는 PBD의 효율성을 높이기 위한 다양한 최적화 기법을 연구하였다[21]. 이 연구는 PBD 알고리즘의 계산 속도를 향상시키기 위해 병렬 처리를 도입하고, 여러 가지 제약 조건을 효율적으로 처리하는 방법을 제안하였다. 이러한 최적화 기법은 PBD가 더 큰 규모의 시뮬레이션에 적용될 수 있도록 하는 데 기여하였다. Kim과 James는 물리 기반 캐릭터 스키닝 기법을 제안하였다[22]. 이 연구는 캐릭터의 물리적 특성을 정확하게 모델링하기 위해 PBD 알고리즘을 활용하였다. 또한, PBD를 이용한 충돌 처리 및 접촉 제약 적용 방법을 제안하여 캐릭터 스키닝의 현실성을 높였다.
본 연구에서 사용된 PBD는 밀도 기반 방법의 표면 검출을 위해 밀도 값을 PBD의 정점에 매핑한다[23, 24]. PBD를 적용하는 시뮬레이션 단위는 단위 크기 육면체인 SimCell로 정의된다[18].
본 연구는 Aguilera가 제안한 병렬 리샘플링을 통해 변형된 볼륨 데이터를 생성, 가시화한다[14]. 재표본화 알고리즘의 볼륨 재표본화 파이프라인은 다음과 같이 구성된다. 전처리 과정에 속한 묵시적 샘플링 메쉬(implicit sampling mesh), 그리고 실행 과정에서 수행되는 볼륨 재표본화(volumetric resampling)이다.
첫 번째 단계에서는 볼륨 데이터의 각 복셀에 대응되는 정점에 데이터를 저장한다. 삼차원으로 서로 인접한 8개의 정점을 이용하여 하나의 육면체를 정의하고, 이 육면체를 분해하면 서로 인접하는 5개의 사면체를 획득할 수 있다. 이와 같은 분해 과정을 5T분해(5T decomposition)라고 한다. 분해된 메쉬는 규칙성을 가져, 볼륨 데이터를 구성하는 수많은 사면체의 정점 좌표를 계산할 수 있다.
샘플링 메쉬의 각 정점에 대한 변형이 물리엔진 등을 이용하여 계산되었다고 가정하면, 원본 데이터에 대한 리샘플링을 수행할 수 있다. 볼륨을 구성하는 모든 사면체에 대하여 변형이 병렬로 처리되며, 각 사면체는 재표본화 과정에 필요한 모든 정보를 포함하고 있다. 각 사면체에 대한 축 정렬 경계 상자(axis-aligned bounding box)를 계산하고, 경계 내에 존재하는 모든 정점은 후보 복셀로 선정된다. 그 다음 모든 후보 복셀에 대한 무게 중심 좌표(barycentric coordinates)를 구하고, 사면체 내에 존재하는 좌표를 가지는 후보 복셀에 대한 변형 연산을 수행한다. 최종적으로 변형된 좌표는 사면체의 네 정점에 저장된 데이터를 사용한 무게 중심의 보간을 통해 복셀에 할당된다. 본 연구는 Aguilera의 방식을 일부 효율적으로 수정하여 구현하였다.
3. 시스템의 구성
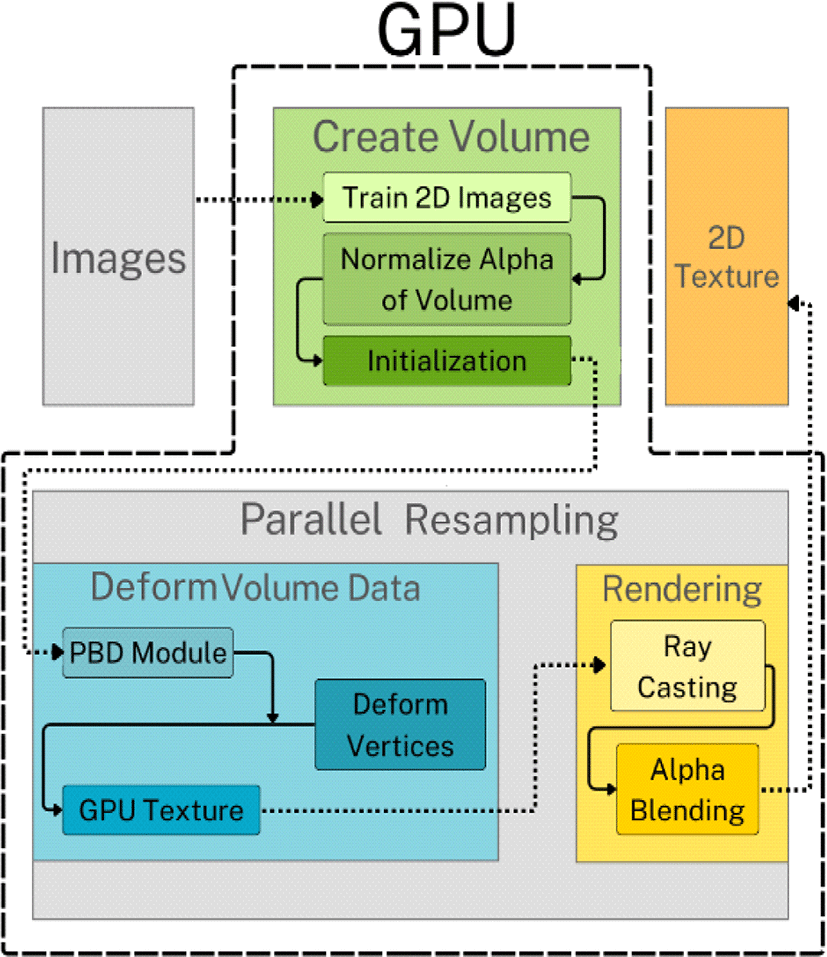
본 연구에서는 NeRF를 이용하여 이차원 영상집합으로부터 삼차원 볼륨 데이터를 생성하고, 이를 실시간으로 변형 및 가시화하는 시스템을 제안한다. 시스템은 크게 세 단계로 각각 볼륨 데이터 생성, 볼륨 데이터 변형, 그리고 렌더링으로 구성된다. 각 단계는 GPU 기반의 고속 처리 및 효율적인 메모리 접근을 통해 실시간 가시화를 가능하게 한다.
NeRF는 색상과 밀도 값을 가진 볼륨 데이터를 생성한다. 볼륨 데이터의 각 복셀은 4가지 채널(r, g, b, a)을 사용하는 색상 값과 밀도 값으로 구성된다. 각 채널은 단일 정밀도(single precision, 4byte)을 사용하여, 복셀 당 16바이트를 필요로 한다. 변형 및 가시화 과정에서 3배의 메모리가 필요한 것을 감안하면, 볼륨 데이터의 해상도 결정에 고려가 필요하다. 예를 들어 의료 데이터에 일반적으로 사용하는 512×512×512 볼륨 데이터 해상도를 가정한다면 볼륨 데이터 생성에만 총 2GB×3=6GB의 메모리가 필요하여 본 제안 시스템의 적용 대상이 제한되는 문제가 있다. 따라서 본 연구에서는 생성하는 볼륨 데이터의 해상도를 256×256×256의 크기로 낮추어 768MB의 메모리를 사용하는 볼륨 데이터를 생성한다.
생성된 원시 볼륨 데이터는 0부터 무한대의 범위를 가지는 밀도 값을 포함하고 있다. 본 연구에서 사용하는 알파 블렌딩 규칙에 적용할 수 있도록 0부터 1 사이의 범위로 밀도 값을 정규화 하는 과정을 수행한다. 이에 대한 구체적인 내용은 본 논문의 4.2절에서 설명한다. 이제 생성된 볼륨 데이터를 입력으로 하는 볼륨 데이터 변형 단계에서 변형 및 보간을 효율적으로 수행할 수 있도록 볼륨 데이터를 GPU의 텍스처 객체(Figure 1. GPU Texture 참고)로 변환하는 과정이 필요하다[25]. 이와 같은 일련의 과정을 통해, NeRF에서 생성된 볼륨 데이터는 GPU 상에서 변형될 준비를 마친다.
본 단계에서는 PBD 모듈에서의 제약 조건들을 활용하여 일정 시간마다 실시간으로 변형된 정점 좌표를 계산하고, 이를 통해 변형된 데이터가 저장된 새로운 볼륨 데이터를 실시간으로 생성한다.
앞 단계인 NeRF에서 생성된 볼륨 데이터는 256×256×256 크기로, 이를 각 정점으로 삼아 PBD로 물리 시뮬레이션을 계산하면 실시간성을 보장하기 어렵다. 따라서 본 연구에서는 물리 시뮬레이션의 해상도를 가시화 해상도보다 낮게 설정하였다. 물리 시뮬레이션의 해상도는 64×64×64로 설정하고 시뮬레이션을 수행한 후, 변형된 이동 벡터를 보간하여 고해상도 가시화 볼륨의 생성에 사용한다.
고해상도 볼륨은 병렬 리샘플링 기법[26]을 변형하여 생성한다. 256×256×256 볼륨 데이터를 인접한 8개의 복셀로 묶어 하나의 셀(cell)로 처리하면, 볼륨 데이터는 255×255×255 개의 육면체 셀로 분해할 수 있다. 각각의 셀을 2.3절에서 설명한 바와 같이 5개의 사면체로 분해하고 각 사면체의 꼭짓점의 평행 이동을 PBD 결과에서 추정할 수 있다. 이렇게 하나의 사면체의 변형이 계산되며, 변형된 사면체 내부의 값을 무게중심 좌표를 이용하여 선형 보간으로 채우는 과정을 반복하면 변형된 전체 볼륨 데이터가 완성된다.
기존 방법[26]은 단일 채널 정수형 (16bit short) 볼륨 데이터를 입력 및 출력 볼륨 데이터로 가정하지만, 본 방법에서는 이미 색상이 부여된 4채널 실수형 (float4) 볼륨 데이터를 입력 및 출력 데이터로 한다. 이에 따라 GPU를 이용한 보간 시 텍스처 샘플링의 설정을 변경해야 한다. 한편, 본 연구의 메모리 사용량이 기존 방법[26]에 비해서 8배 증가하였지만, 상대적으로 데이터 해상도도 512×512×512에서 256×256×256으로 8배 감소하여 실시간성을 보장할 수 있다.
본 단계에서는 이전 단계에서 변형된 볼륨 데이터를 가시화한다. 광선 투사(ray casting) 기법[27]을 사용하여 볼륨 데이터를 이차원 영상으로 가시화하고, 이를 화면에 출력한다.
앞 절의 볼륨 데이터 변형 과정에서 생성되는 변형된 볼륨 데이터는 GPU에서 저장하기 편리한 선형 주소로 구성되어 있다. 따라서 광선 투사 과정에서 유클리드 공간에서는 인접하나 선형 메모리에서 멀리 떨어진 메모리들을 반복적으로 샘플링 할 때 효율이 저하되는 문제가 있다. 이에 따라 본 연구는 볼륨 데이터를 GPU 텍스처로 변형하여 하드웨어 보간, 텍스처 캐시, 자동 경계 검사를 활용할 수 있도록 하였다.
이 과정은 GPU 메모리에서 하나의 볼륨 데이터를 더 생성해야 하고, GPU 메모리 사이의 복사가 한 번 수행되는 비용이 추가된다. 그러나 GPU 메모리의 사이의 복사 속도는 초당 700GB 정도로 알려져 있어 복사 시간은 1ms 정도로 실시간 가시화에 영향을 미치지 않는다. 게다가 가시화 과정에서 근접 좌표에 대한 삼선형 보간이 매우 많이 수행되므로 메모리를 한 번 복사하는 비용에 대비하여 GPU 텍스처 사용의 이득이 크다고 판단하였다.
4. 시스템 개발 고려사항
NeRF의 특성상 생성된 원시 볼륨 데이터의 중심 영역은 정확한 데이터가 생성된다. 반면에, 중심으로부터 멀어질수록 노이즈가 심한 결과가 자주 관찰된다. 이는 관찰 카메라가 원본 데이터 모델의 중앙 부분에 많은 시간을 들여 영상을 집중적으로 촬영하기 때문이다. 이러한 원시 볼륨 데이터를 그대로 사용하면 노이즈 및 먼지 입자 등의 낮은 밀도 값을 가지는 영역 때문에 실제 물체 형상을 정확히 반영하지 못하는 문제가 발생한다.
본 연구에서는 이러한 문제를 해결하기 위해 전처리과정을 수행한다. 먼저, 볼륨 데이터의 중심점과 각 축(x, y, z)을 방향으로 설정한 반경을 기준으로 하는 타원체(ellipsoid)를 정의한다. 각 픽셀의 좌표와 중심점 사이의 거리를 이용해 각 복셀에 대한 타원체 내부의 포함 여부를 판별한다. 이때, 타원체 외부 영역의 복셀들은 밀도 값을 0으로 설정한다. 또한, NeRF 모델에서 출력되는 밀도 값에 대해 임계값(threshold)을 적용하여, 해당 임계값보다 낮은 밀도 값을 가지는 영역은 완전히 투명하게 처리한다. 결과적으로 원시 볼륨 데이터에서 다루려는 물체 주변에 발생되는 노이즈 및 먼지 입자들을 제거하여 올바른 변형과 가시화를 수행할 수 있다.
본 연구에서는 NeRF로부터 생성된 볼륨 데이터의 밀도 값을 기존의 알파 블렌딩(alpha blending) 기법에 맞게 정규화 하였다. 먼저 NeRF에서 사용하는 밀도 값을 0에서 1 사이의 불투명도 값으로 변환하는 과정에서 아래와 같은 정규화 식을 사용한다.
이때 알파(α)는 0 ~ 1 사이의 정규화 된 불투명도 값, 타우(τ)는 정규화 전 가상 입자의 밀도 값으로 0에서 무한대의 값을 갖게 된다. 알파 정규화는 NeRF에서 추출한 밀도 값을 본 연구의 알파 블렌딩 규칙에 맞추기 위해 중요하다.
본 연구에서는 NeRF를 사용하여 생성된 볼륨 데이터에 대한 물리 기반 시뮬레이션을 적용하였다. 기존의 CT 촬영이나 MRI 검사와 같은 밀도 기반 광선 촬영 기법에서는 인체의 외부와 내부 밀도 값이 모두 포함되어 있었으나, NeRF에서 제공되는 볼륨 데이터의 경우 표면 밀도 정보만 학습되어 전달되는 한계가 있다.
기존 PBD 알고리즘에서는 CT나 MRI 데이터에 저장된 밀도 정보를 통해 물체의 강성(stiffness)을 결정한다. PBD에서 강성 값은 물체가 변형에 저항하는 정도를 나타내며, 높은 강성 값을 사용할 경우, 물체는 더 단단해지고 변형이 적어지는 반면, 낮은 강성 값을 사용할 경우 물체는 더 유연해지고 쉽게 변형된다[20].
하지만 CT나 MRI에서 얻은 밀도 정보는 내부 데이터까지 정확하게 포함하고 있어 시뮬레이션 결과가 상대적으로 정확한 반면, NeRF 볼륨 데이터는 내부 밀도 정보를 정확히 제공하지 못한다. 이로 인해 PBD에서 일반적으로 간선에 강성을 결정할 때 밀도 값의 평균으로 변형체의 강성을 결정하는 방식을 그대로 적용하면, 물체가 지나치게 출렁이는 효과가 발생할 수 있다. 이러한 문제를 해결하기 위한 방법으로, 본 연구에서는 NeRF에서 제공되는 밀도 값을 활용하여 PBD 물리엔진에 적용하되, 강성 값을 사용자 지정 파라미터로 설정하여 직접 보정할 수 있게 하였다.
본 단락에서는 변형된 데이터를 이차원 텍스처로 변환하여 화면에 출력하는 과정을 설명한다. 가시화는 광선 투사 기법[27]을 통해 삼차원 볼륨 데이터를 이차원 영상으로 변환한다. 광선은 볼륨 데이터를 통과하면서 각 위치에 대한 색상 값과 밀도 값을 통합하여 최종 이차원 영상을 생성한다. 본 시스템에서 생성한 볼륨 데이터에는 색상 값과 밀도 값이 저장되어 있으므로, 분류(classification)작업은 생략된다. 또한, 본 연구는 조명 연산도 포함하지 않았다. 이는 법선벡터의 추정이 노이즈에 의해 원활하지 않았기 때문이다.
GPU를 이용한 볼륨 가시화에서 각 좌표의 샘플링 과정에서는 RGBA가 저장된 볼륨 데이터에서 선형 보간을 수행한다. 한편, float 정밀도를 가진 4채널의 볼륨 데이터에서 삼차원 선형 보간을 수행하려면 해당 하드웨어에서 기능을 지원해야 한다. 이렇게 획득한 샘플 값을 각 픽셀에 대해 알파 블렌딩으로 누적하였다. 일반적인 광선 투사법과 동일하게 조기 광선 종료(early ray termination) 가속 기법을 적용하였다.
Input: rayStartPosition, volumeData
Output: Accumulated Color in Image
if CalculateRayBoundary(rayStartPosition, volumeData) ≠0 then
Calculate intersection points with AABB
Determine intersection points before and after transformation
else
if rayStartPosition is outside volume range then
return
end if
end if
for each point from initial to final intersection point do
samplingPoint ← sample along new ray
newColor, alpha ← ExtractColorAlpha(samplingPoint)
accumulatedColor ←
accumulatedColor + alpha × newColor ×
(1 - accumulatedAlpha)
accumulatedAlpha ← accumulatedAlpha + alpha ×
(1 - accumulatedAlpha)
if accumulatedAlpha > 0.99 then
return
end if
end for
StoreColorInImage(image, accumulatedColor)
5. 실험
본 연구의 실험은 AMD Ryzen 9 3900X 12-Core Processor 3800MHz CPU, 64GB 메모리를 가지며, NVIDIA Geforce RTX 3090 GPU를 사용하여 수행되었다. 소프트웨어 구현은 Visual Studio C++를 사용하였으며, 딥러닝 알고리즘은 Instant-NGP(Instant Neural Graphics Primitives)[28]을 통해 가시화에 사용된 학습은 대략 2500 – 3000 스텝(step)을 수행한다.
사용한 메모리 정보는 Table 1과 같다. 삼차원 텍스처의 크기는 2의 지수형태만 가능하며, 512×512×512의 데이터(2GB)로 실험해본 결과, 256×256×256 데이터에 비해 데이터 변형 시간이 약 43배, 렌더링 시간이 약 26배 더 소요된다. 이는 메모리 접근에서 많은 병목현상이 생기는 것으로 추정된다. 이에 512×512×512 크기의 볼륨 데이터는 실험으로 사용하지 않았다. Volume에는 NeRF에서 생성된 각 복셀이 4개의 채널을 가진 볼륨 데이터가 저장되며, texture에는 볼륨 데이터를 이차원 텍스처화 한 색상 정보를 저장한다. 동영상은 iPhone 14 Pro 스마트폰의 기본 카메라 애플리케이션을 사용하여 촬영되었으며, 1분 20초 동안 사람의 얼굴을 다양한 각도에서 촬영하여 전체적인 모습이 영상에 표현되도록 하였다.
| Dimension | Channel | Size | |
|---|---|---|---|
| Volume | 256x256x256 | 4 | 256MB |
| Texture | 512x512 | 3 | 768KB |
NeRF 모델에서 출력되는 볼륨 데이터는 각 위치의 밀도 정보를 제공한다. 이 밀도 값은 0부터 무한대의 밀도 값을 가진다.
Figure 2는 임계값(threshold) 처리 전후의 볼륨 데이터를 비교한 결과를 나타낸다. (a)의 결과를 확인하면 낮은 밀도 영역이 그대로 표현되어 실제 물체의 형상이 드러나지 않는다. 반면 (b)의 경우 형상이 드러나는 것을 볼 수 있다.

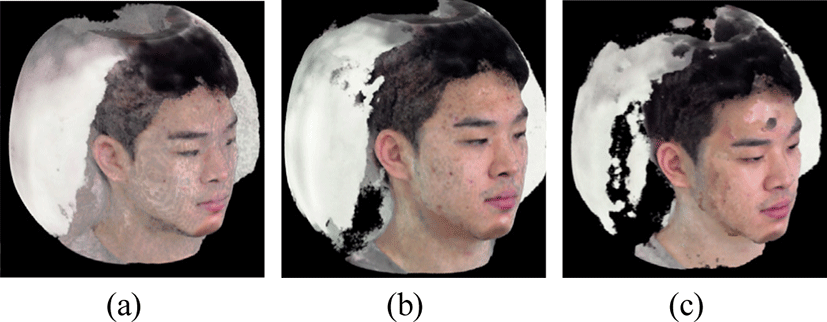
Figure 3은 볼륨 데이터의 임계값을 5, 20, 35로 늘렸을 때의 추가 실험 결과를 나타낸다. 영상 (a)의 결과는 낮은 임계값으로 인해 미세한 먼지 입자들까지 많이 표현된다. 물체의 전체적인 형상은 잘 나타나지만, 표면이 다소 거칠고 불규칙한 모습을 보인다. 그에 비해 영상 (b)에서는 본 연구에서 사용한 기본 임계값으로, 물체의 전반적인 형상과 표면이 비교적 자연스럽게 표현되는 모습이 확인된다. 미세한 먼지 입자들이 제거되므로 영상(a) 와 비교하였을 때, 보다 깨끗한 결과를 보인다. 또한, 물체 내부의 세부 구조도 적절히 드러나는 균형 잡힌 결과를 나타낸다. 영상 (c)의 결과는 높은 임계값으로 인해 물체 표면의 일부가 누락되거나 구멍이 생성되는 문제가 발생한다. 물체 내부의 세부 구조는 잘 드러나지만, 전체적인 형상이 왜곡되어 있다.

실험에 사용된 데이터 외에도 동일한 파라미터 값을 다양한 데이터에 적용한 결과, Figure 4와 같이 비슷한 변화 양상을 보인다. 이는 파라미터들이 특정 데이터에 종속되지 않고 일관되게 적용될 수 있음을 확인하고, 파라미터 설정이 다양한 상황에서도 신뢰할 수 있는 결과를 제공함을 의미한다.
이와 같은 실험을 통해, 본 연구에서 사용한 임계값이 20 정도로 반영되었을 때, NeRF가 생성한 볼륨 데이터의 특성이 가장 잘 반영되는 것으로 판단한다. 이 값은 물체의 전반적인 형상과 세부 구조를 적절히 표현하면서도 불필요한 먼지를 효과적으로 제거할 수 있다.
0부터 무한대의 값을 가지는 밀도 값(5.1)의 정규화 효과를 Figure 5에 제시하였다. 이처럼 밀도 값을 정규화 하는 것은 NeRF 볼륨 데이터의 RGB 색상을 더욱 정확하고 현실적으로 표현하는 데 매우 중요하다. 원시 밀도 값을 그대로 사용하면 색상이 왜곡되어 보이지만, 후보정을 수행한 과정을 거치면 물체의 실제 색상과 비슷하게 구현되는 모습이 Figure 4에서 확인된다. 이러한 정규화 된 밀도 값은 NeRF를 활용한 삼차원 시각화의 정확성과 효율성을 높인다.

본 연구는 다양한 강성 값을 사용한 결과들을 비교하였다. 실험 결과, 강성 값을 1.0 이하로 설정할 경우 내부 볼륨 데이터가 과도한 출렁거림을 일으키는 문제가 발생한다. 이에 따라 강성 값을 1.0으로 고정하는 것이 적절하다고 판단하였다. 이 실험은 변형체 해상도 64×64×64을 사용하여 수행되었다.
Figure 6은 실험 결과를 가시화한 영상이다. 영상 (a, c)는 강성 값이 1.0으로 설정된 경우를 나타내며, 영상 (b, d)는 강성 값이 0.8로 설정된 경우를 나타낸다. 강성 값이 1.0인 경우가 더 안정적인 볼륨 변형 결과를 제공하며, 출렁거림 현상이 최소화된 결과를 확인할 수 있다.
이와 같은 연구 결과는 NeRF 데이터의 특성을 고려하여 PBD 알고리즘에 적절한 강성 값을 대응시킴으로써, 표면 정보만 제공되는 볼륨 데이터에 대해서도 물리 기반 시뮬레이션을 수행할 수 있음을 보인다.
PBD(물리 기반 변형) 외에도, 사전 계산된 간단한 공식을 사용하여 볼륨 데이터를 변환할 수 있다. 다양한 변형 규칙을 적용함으로써 볼륨 데이터를 다양한 시각적 형태로 변환할 수 있다. 수식 기반 변형은 특정 축이나 중심점을 기준으로 다양한 변형을 자유롭게 적용할 수 있어, 특정 분석 목적에 따라 데이터를 맞춤형으로 변형할 수 있다.
다음은 간단한 변환 규칙을 수식으로 생성하여 볼륨 데이터를 변환한 결과이다. 각 변환은 특정 축이나 중심점을 기준으로 회전, 이동 및 크기변환을 수행한 것으로 결과는 Figure 7에서 확인된다.
영상 (a)에서는 z축 좌표와 현재 시간을 결합하고, sin 함수에 적용하여 y축 방향으로 주기적인 세로 이동을 적용한다. 이 변환은 주로 y축을 중심으로 이루어진다. 영상 (b)에서는 z축을 중심으로 회전한다. 중심점과의 거리 및 시간에 따라 회전 각도를 계산하여, 점을 중심으로 회전시킨다. 마지막으로 영상 (c)에서는 중심점에서의 거리와 시간에 따라 점을 구형으로 변형시킨다. 이 변환은 중심점을 기준으로 방사형으로 확장과 수축을 적용하며, 변위 벡터를 계산하여 크기를 조정한 후, 중심점으로부터의 변형된 위치를 구한다.
이와 같이, 사전 계산된 간단한 공식을 사용하여 다양한 형태의 변환을 적용할 수 있다. 본 연구에서는 물리엔진을 적용한 변형을 포함하여 볼륨 데이터의 기하학적 변형을 효과적으로 수행할 수 있다는 결과를 도출했다.
6. 결론
본 연구는 NeRF와 PBD, 병렬 리샘플링을 결합하여 실시간으로 삼차원 볼륨 데이터를 생성, 변형 및 시각화하는 시스템을 제안하였다. NeRF는 이차원 영상과 카메라 포즈 정보를 입력으로 사용하여 고해상도 삼차원 볼륨 데이터를 생성한다. PBD는 물리 기반 시뮬레이션 기법을 이용하여 실시간 변형과 상호작용을 가능하게 한다. 병렬 리샘플링을 통해 볼륨을 작은 사면체 메쉬들로 분할하고 변형된 볼륨 데이터를 생성한다. 이후 볼륨 가시화를 수행한다.
NeRF는 데이터 중 일부를 학습하여 새로운 시점에서의 영상을 생성(inference)하고, 나머지 데이터(ground truth)와 비교하여 생성한 영상과의 손실(loss)를 비교해 역전파(back propagation)로 학습을 진행한다. 반면, 본 연구에서는 새로운 시점에서 생성한 이미지를 바로 변형시키기 때문에 화질이 저하되는 한계가 있다. 그럼에도 불구하고, 본 연구는 실시간 변형과 빠른 연산 속도를 제공하는 장점이 있다.
특히, 본 연구의 차별점은 볼륨 데이터 변형 과정에서 물리엔진을 활용한다는 것이다. 이는 실시간으로 변형된 데이터를 가시화할 수 있게 하며, 물리적 제약 조건을 적용하여 보다 다양한 시뮬레이션을 가능하게 한다. 우리의 목표는 정적인 NeRF 렌더링 화면에서 동적인 물리엔진 기반의 프로젝트를 결합하는 것이었으며, 이를 통해 실시간 변형과 가시화를 실현할 수 있음을 보였다.
본 연구의 또 다른 한계로서, 본 연구의 주된 목표는 통합된 시스템 설계였기 때문에 이론적 배경을 구축하는 데 부족함이 있었다. 향후, 생성된 볼륨 데이터에서 노이즈를 효과적으로 제거하는 방법이나, PBD 물리 계산에서 강성 값을 자동으로 보정하는 방법 등을 연구할 수 있다.
본 연구는 NeRF, PBD, 병렬 리샘플링을 결합한 실시간 삼차원 볼륨 데이터 생성, 변형 및 시각화 시스템을 시험적으로 구현하였다. 이 시스템은 공학, 교육, 의료 등 다양한 분야에서 활용될 수 있는 잠재력을 가지고 있으며, 치아나 모발과 같은 간단한 교정 시술 예측에서 사용될 수 있을 것으로 기대한다.