1. 서론
전립선암(prostate cancer, PCa)은 전 세계 남성에서 발생 빈도가 두 번째로 높은 암이며, 암으로 인한 사망 원인 중에서도 다섯 번째로 높아 매우 위험한 암이다[1]. 환자의 적절한 치료 계획을 수립하고 예후를 관리하기 위해서는 전립선암의 악성도를 조기에 예측하는 것이 필요하다[2]. 현재 전립선암의 악성도 측정을 위한 일반적인 방법은 조직의 일부를 채취하는 조직 생검을 수행하여 가장 많이 나타나는 암 세포 패턴과 두번째로 흔하게 발견되는 암 세포 패턴에 대해 1~5의 등급을 부여하고 이를 합산하는 글리슨 점수 방법이다[3]. 이 때, 글리슨 점수가 높으면 전립선암의 악성도가 높음을 의미하여 고위험군(high grade PCa, hPCa)에 속하고, 반대로 글리슨 점수가 낮으면 전립선암의 악성도가 낮음을 의미하여 저위험군(low grade PCa, lPCa)에 속한다. 저위험군으로 예측된 환자에게는 추적 관찰을 통해 과잉 진단의 위험과 불필요한 치료로 인한 부작용을 줄일 수 있고, 고위험군으로 예측된 환자에게는 적절한 치료 계획을 수립하여 환자의 예후를 개선할 수 있기 때문에 전립선암 악성도의 정확한 조기 예측이 필요하다. 그러나 조직 생검 작업은 바늘을 찔러 넣는 침습적 방식으로 출혈에 의한 패혈증과 합병증의 위험이 있어, 이를 줄이기 위해 자기공명영상(Magnetic Resonance Imaging, MRI)을 활용한 딥러닝 모델 기반의 비침습적 전립선암 악성도 예측 방안이 시도되고 있다.
전립선암 악성도 예측을 위한 딥러닝 모델은 의사가 입력 데이터에 지정한 분류 정답 값을 기준으로 학습하는 지도학습 방식을 일반적으로 사용한다. 이 때, 입력 영상으로는 T2강조MR영상 (T2-weighted MR Imaging, T2wMR), 확산 강조 영상(diffusion-weighted imaging, DWI), 현상 확산 계수 지도(apparent diffusion coefficient map, ADCmap) 등의 다중 파라미터 MR 영상이 있으며, 해당 영상들의 특징을 전립선암 악성도 예측에 활용하는 방안이 제안되었다. Aldoj등[5]은 공공 데이터셋 ProstateX의 T2wMR, DWI, ADCmap, k-trans 영상을 다양한 조합으로 3채널 영상을 획득해 글리슨 점수 7점 이상의 고위험군을 예측하는 CNN 모델에 적용했으며, DWI, ADC, K-trans 영상 조합에서 AUROC(Area Under the ROC Curve) 점수 0.897로 가장 좋은 성능을 보였다. Sanford등[6]은 ProstateX 데이터셋과 병원 데이터셋의 T2wMR, DWI, ADCmap을 종양을 중심으로 크롭(crop)한 후 3채널로 합쳐 만든 영상을 사용하여 ResNet34네트워크로 PIRADS 점수 2점 이상의 고위험군을 예측할 방법을 제안하였고, AUROC 점수 0.726을 보였다. 그러나, 이러한 연구들은 모두 지도학습 방식을 사용하고 있기 때문에 의사가 분류 정답 값에 해당하는 레이블을 지정하는 데 많은 시간과 비용이 소요되어 충분한 데이터셋을 얻기 어렵다는 한계가 있으며, 훈련에 사용되는 데이터셋이 적을수록 모델의 예측 성능이 떨어질 수 있다.
이를 극복하기 위한 방안으로 자기지도학습(self-supervised learning)[8]이 주목받고 있다. 자기지도학습은 레이블이 없는 데이터셋으로부터 영상 정보의 패턴을 학습하는 방식으로, 의료 분야와 같이 데이터 수집이 제한적인 환경에서 효과적으로 영상의 특징을 추출하기 위해 다양하게 활용되고 있다. Li 등[9]은 CNN과 자기지도학습 방식으로 훈련된 오토인코더의 이중 입력값으로 T2wMR과 DWI를 사용하고, 채널 및 위치 어텐션을 수행하여 글리슨 점수 7점 이상의 고위험군에 대해 AUROC 점수 0.84의 예측 성능을 도출했다. Rossi 등[10]은 T2wMR과 DWI의 입력 영상과의 시각적 유사도를 평균제곱오차로 계산하여 가장 유사한 영상들을 일정 개수만큼 선별한 후 이들 중 가장 많이 나타나는 클래스로 입력 영상을 분류하는 방식을 제안했으며, PIRADS 점수 4점 이상의 고위험군에 대해 T2wMR에서는AUROC 점수 0.69, DWI에서는AUROC 점수 0.76의 결과를 보였다. Shin 등[11]은 SimCLR 프레임워크 기반의 자기지도학습 모델을 ProstateX 공공 데이터셋으로 추가 학습시킨 후 T2wMR, DWI, ADC지도 영상에서의 전립선암 악성도 예측값을 투표 방식으로 결합하는 방안을 제안했으며, 글리슨 점수 4+3점 이상의 고위험군에 대해 AUROC 점수 0.76의 성능을 보였다. 기존 연구들은 전립선암 악성도 예측값 도출에 다중 파라미터 MR 영상들의 영상 정보를 반영하고자 전립선암 악성도 예측 과정에서 이를 융합하는 형태를 보인다. 그러나 전립선 MR 영상을 대상으로 하여 전립선암 악성도를 예측하는 연구들 중 자기지도학습 과정에서 다중 파라미터의 MR 영상 정보를 종합하여 인코더를 학습시키는 연구 사례는 많지 않다.
따라서 레이블 데이터의 확보가 어려운 의료 분야에서의 한계점을 극복하고 다중 파라미터 MR 영상들의 활용을 극대화하기 위한 방법으로, 자기지도학습 과정에서 다중 파라미터 MR 영상 및 특징들을 융합하여 전립선암 악성도 예측을 개선하는 방법을 제안한다. 본 연구의 독창성은 세가지로 요약할 수 있다. 첫째, 자기지도학습 과정에서 다중 파라미터 MR 영상들을 사용하여 여러 영상의 특징을 함께 추출하고 이를 융합한다. 둘째, 전립선암 악성도 예측 모델에 필요한 주요한 특징을 더 잘 학습하기 위해, 자기지도학습 과정에서 전립선암 악성도 예측에 사용될 데이터셋으로 모델을 추가 학습한다. 셋째, 다중 파라미터 MR 영상들의 융합과 자기지도학습 모델에 대한 추가 학습이 전립선암 악성도 예측 정확도 향상에 미치는 영향을 확인하기 위해, 단일 파라미터 MR 영상만을 활용했을 때와의 예측 정확도를 비교하고 추가 학습 전후 전립선암 악성도 예측 결과의 t-SNE 분포 변화를 분석한다.
2. 제안 방법
전립선암 악성도 예측을 위한 제안 방법은 두가지 과정으로 이루어진다. SimCLR 프레임워크를 사용한 자기지도학습 과정과 자기지도학습 모델의 인코더를 활용해 실제 전립선암 악성도 예측을 수행하는 과정이다.
전립선암 악성도 진단을 위해 사용되는 다중 파라미터 MR 영상에는T2wMR, DWI, ADCmap이 있다. T2wMR은 고화질의 장점이 있지만, 조직검사로 인한 출혈이 있을 경우 출혈 부위에 낮은 밝기값을 보여 정확한 판독을 저해할 수 있다는 단점이 있다. DWI와 ADCmap은 전립선암과 주변 조직 간의 신호 강도 대비가 크게 나타나 종양 부위 관측에 유리하지만 낮은 해상도로 인해 단독으로 사용하기 어렵다는 한계가 있다. 이러한 단일 파라미터 MR 영상들의 장단점을 상호보완하기 위해 다중 파라미터 MR 영상의 다양한 영상 특징을 종합적으로 제공함으로써 자기지도학습 모델이 전립선암 종양 영상의 특징을 더욱 효과적으로 추출할 수 있도록 한다. 따라서 자기지도학습 과정에서 다중 파라미터 MR 영상을 융합해 사용하여 전립선암 악성도 예측에 유용한 특징들을 추출하고자 한다.
본 연구에서는 자기지도학습의 기본 프레임워크로서 SimCLR[12]를 사용한다. SimCLR 은 대조적 학습 방법으로, 자기지도학습을 수행하는 대표적인 방식이다. 각 입력 영상에 대해 데이터 증강을 통해 2개의 변환된 영상을 생성하고, 같은 영상에서 변환된 영상 특징 간의 유사도는 최대화하며, 다른 영상에서 변환된 영상 특징 간의 유사도는 최소화하는 방식으로 학습을 진행한다. SimCLR의 네트워크는 영상의 특징을 추출하는 인코더와 추출한 특징을 특정 차원의 벡터로 축소하기 위한 프로젝션 헤드(Projection Head)로 구성된다. 인코더는 ResNet18[13]을 사용하며, 프로젝션 헤드는 두 개의 MLP(Multi-Layer Perceptron)와 이들 사이의 ReLU(Rectified Linear Unit) 활성화 함수로 구성된다. 대조적 학습 방법의 식 (1) 과 (2)는 대조 손실 함수와 축소된 특징 벡터 간의 거리를 코사인 유사도로 표현한 식을 의미한다.
이 때, zi와 zj는 입력 영상이 자기지도학습 프레임워크를 거쳐 나온 출력 벡터이며, si,j는 이들 간의 코사인 유사도를 구한 것이고, N 은 배치 크기를 의미한다. τ 는 유사도의 확률 분포를 조정하여 샘플 간 거리에 가중치를 부여하는 하이퍼파라미터로 본 연구에서는 0.07로 지정한다.
다중 파라미터 MR 영상들의 정보를 자기지도학습 과정에서 반영하여 학습하기 위해 두 가지 융합 방식을 사용한다. 첫 번째, 입력 단계에서의 융합 방식인 영상 융합 방식은 그림 1(a) 와 같이 단일 파라미터 MR 영상을 3채널 색상 영상으로 융합하는 것이다. 다중 파라미터의 MR영상들은 모두 1채널 데이터로, 이를 T2wMR, DWI, ADCmap 순서로 배치하여 하나의 3채널 입력 영상을 생성한다. 이때 각 영상은 동일 환자의 동일 슬라이스 번호에 해당하는 영상들로 구성한다. 두 번째, 단일 파라미터 MR 영상에서 추출한 특징을 융합하는 방식인 특징 융합 방식은 그림 1(b) 와 같이 세가지 파라미터 MR 영상을 각각의 자기지도학습 모델의 인코더의 출력 벡터 차원에서 융합하며, 이로 인해 프로젝션 헤드의 크기도 3배 증가된다.
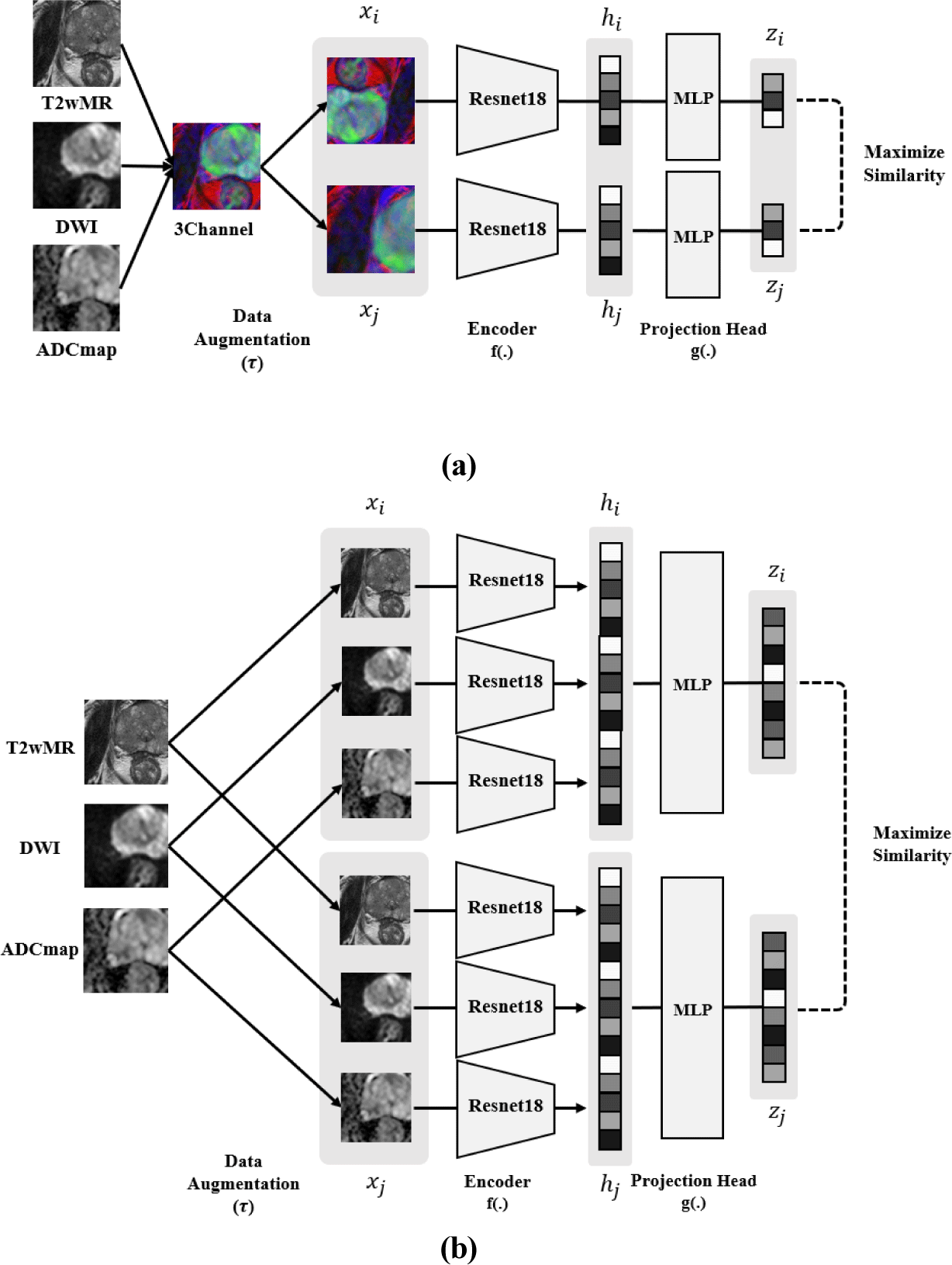
자기지도학습 과정에 적용되는 두 가지 융합 방식은 각각 다른 장점을 제공한다. 영상 융합 방식은 전립선암 부위에 대한 단일 파라미터 MR 영상들 간의 영상 특징 차이를 채널 단위로 추출함으로써 이를 초기에 모델 학습에 반영할 수 있다는 장점이 있다. 특징 융합 방식은 세가지 파라미터 MR 영상 각각의 인코더로부터 추출된 특징을 차원 축소시키는 과정에서 유의미한 정보를 선별적으로 결합함으로써 전립선암 악성도 예측에 중요한 특징을 추출하는 데 유리하다.
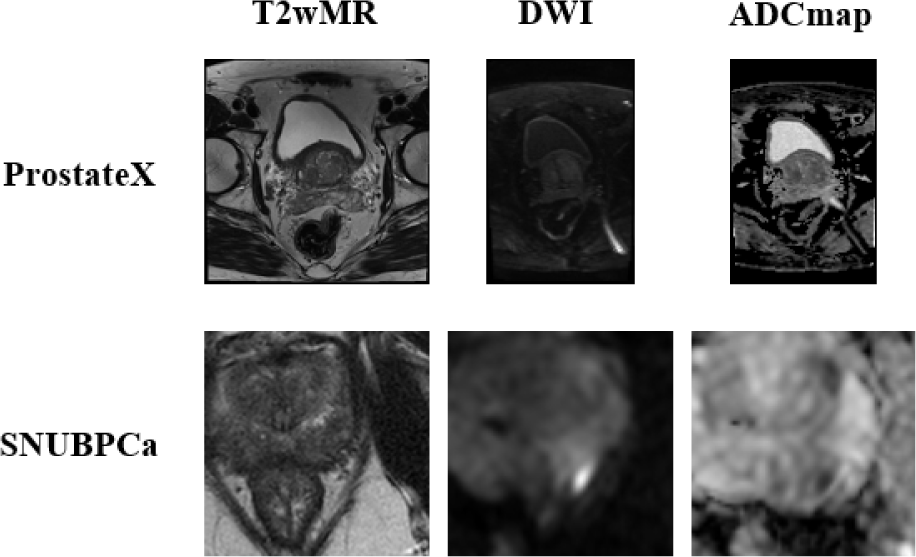
딥러닝 기반 전립선암 악성도 예측 모델 개발에서 학습에 사용되는 데이터셋의 영상 특징은 모델 성능에 영향을 미친다. 전립선암 악성도를 예측할 데이터셋과 유사한 영상 특징을 가진 데이터셋을 모델 훈련 과정에 사용할 경우, 전립선암 악성도 예측에 유효한 영상 특징을 효율적으로 추출할 수 있어 예측 정확도 향상에 기여한다[11]. 그림 2는 본 연구에서 사용할 공공 의료 데이터셋인 ProstateX와 전립선암 악성도를 예측할 데이터셋 SNUBPCa의 영상 특징을 보여준다. 두 데이터셋은 모두 전립선암 영상을 포함하지만, ProstateX는 전립선 전체 영역을 포함하는 반면 SNUBPCa는 종양 부위를 중심으로 일정 크기만큼 크롭되어 서로 다른 영상 특징을 가진다. 또한 DWI의 경우, b-value값이 ProstateX와 SNUBPCa에서 각각 800 s/mm2과 1000 s/mm2인 영상으로 구성되어 있어 두 데이터셋 간의 밝기값에도 차이가 있다. 따라서 본 연구에서는 실제 예측에 사용할 데이터를 사용하여 자기지도학습을 추가 수행한 모델을 사용한다. 전립선암 악성도 예측 네트워크는 자기지도학습 모델의 인코더 뒤에 새로운 완전연결계층(fully connected layer)을 추가하여
구성되며, 예측 네트워크 학습을 통해 네트워크의 전체 계층을 추가로 미세 조정한다. 각 단일 파라미터 MR 영상들에 해당하는 세가지 전립선암 예측 네트워크들은 모두 자기지도학습 과정에서 사용되었던 영상과 동일한 파라미터의 MR 영상 하나를 입력받아 하나의 예측값을 도출한다. 영상 융합 방식과 특징 융합 방식의 경우는 자기지도학습 과정에서와 동일한 형태로 입력값을 받도록 전립선암 악성도 예측 네트워크가 구성되기 때문에, 영상 융합 방식에서는 예측 네트워크가 3채널로 융합된 영상 하나를 입력 받고, 특징 융합 방식에서는 각각의 단일 파라미터 MR 영상을 하나씩 입력 받음으로써 최종적으로는 두 방식의 예측 네트워크 모두 하나의 예측값을 도출한다.
전립선암 악성도 예측 과정에서 수행되는 융합 방식은 예측 융합 방식으로, 단일 파라미터 MR 영상들에 해당하는 각 네트워크의 예측 값을 사용하여 전립선암의 고위험군 여부를 산정하는 방식으로 수행된다. 예측 융합 방식은 그림 3과 같이 과반수 투표(Majority Voting, MV) 방식과 평균 투표(Average Voting, AV) 방식이 있다. 과반수 투표 방식은 단일 파라미터 MR 영상의 모델들 중 과반수 이상이 예측한 값으로 최종 클래스를 결정하며, 평균 투표 방식은 각 모델의 분류 결과의 확률을 평균 내고 가장 높은 평균값을 가진 클래스를 최종 클래스로 선택하는 방식이다.
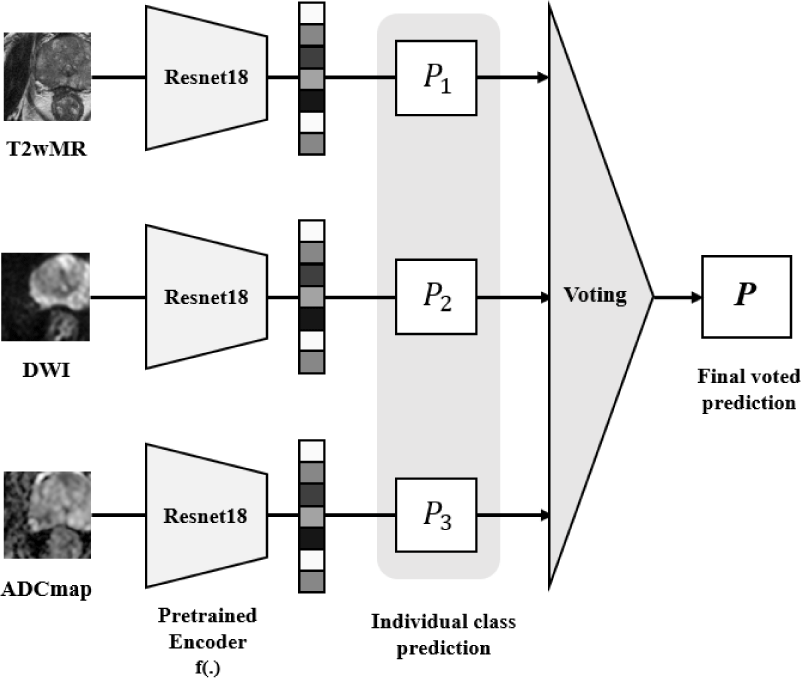
예측 융합 방식은 하나의 예측 네트워크가 주요 특징을 포착하지 못해 잘못 분류하더라도 다른 네트워크들의 결과를 함께 반영하고 상호 보완함으로써 더 정확하고 포괄적인 전립선암 악성도 예측이 가능하다는 장점이 있다.
3. 실험 및 결과
실험에 사용한 데이터셋은 ProstateX[14]와 SNUBPCa 데이터셋 두 가지이다. ProstateX는 전립선암의 다중 파라미터 MR 영상들로 구성된 공공 데이터셋으로, Siemens 3T MR 기기로 촬영한 309,251개의 데이터를 포함하고 있다. ProstateX 데이터셋 중 터보 스핀 에코 시퀸스로 획득한 T2wMR, DWI, ADCmap을 사용한다. SNUBPCa은 분당서울대병원에서 임상시험 윤리위원회(IRB)의 승인을 받은 데이터셋으로, 314개의 종양 영상을 포함하고 있다. 이 영상은 전립선 특이항원검사 (Prostate Specific Antigen test, PSA test)와 경직장 초음파 및 직장 수지 검사를 통해 전립선암으로 진단된 환자 249명으로부터 획득되었다. 이 데이터셋은 3.0T MR 영상 스캐너 (Philips Achieva) 로 촬영되었으며, DWI는 b-value가 1000 s/mm2인 영상을 사용하였고, ADCmap은 b-value 0 s/mm2과 1000 s/mm2의 DWI영상들로부터 획득되었다. 총 314개의 종양 데이터 중 174개의 저위험군 데이터는 글리슨 점수 3+3인 8개, 3+4인 166개로 구성되어 있으며, 140개의 고위험군 데이터는 글리슨 점수 4+3인 100개, 4+4인 16개, 4+5인 21개, 5+4인 3개로 구성되어 있다. 또한 자기지도학습 과정에서는 ProstateX 데이터셋과 SNUBPCa 데이터셋을 모두 사용하였고, 전립선암 악성도 예측 과정에서의 미세조정에는 SNUBPCa 데이터셋을 훈련 데이터셋 189개, 검증 데이터 72개, 테스트 데이터셋 52개로 나누어 사용했다. SNUBPCa 데이터셋은 종양을 중심으로 일정 크기로 자른 패치를 생성한 후, 최소-최대 정규화 (min-max normalization)를 수행하여 환자 간 밝기값을 일치시킨 후, 시간차로 발생하는 움직임을 보정하기 위해 다중 파라미터 MR 영상 간의 강체 정합을 수행하였다.
제안 방법에 대한 실험은 Python 언어와 Pytorch 프레임 워크를 활용하여 진행되었다. 자기지도학습 과정과 전립선암 악성도 예측 과정 모두 NVIDIA GeForce GTX1080Ti Black ScaleD5X 11GB 4개가 장착된 서버에서 수행되었다. ImageNet의 가중치를 바탕으로 ProstateX 데이터셋을 사용해 전이 학습을 하는 과정에서는 배치 사이즈 (batch size)를 128, 학습률 (learning rate)을 5e-5, 가중치 감쇠 (weight decay)를 1e-4로 설정하고, 인내도는 200번으로 설정하였다. ProstateX로 전이 학습을 마친 모델을 기반으로 SNUBPCa데이터셋을 사용해 추가 전이 학습을 할 때는, 배치 사이즈 128, 학습률 4e-4, 가중치 감쇠 1e-4, 인내도 200으로 하이퍼파라미터를 설정하였다.
제안된 방법의 결과 분석을 위해, 단일 파라미터 MR 영상들에서 전립선암 악성도를 예측한 결과와 영상 융합, 특징 융합, 예측 융합을 수행했을 때의 예측 결과를 비교하여 제시한다. 성능 평가를 위한 정량적[15] 지표로는 정확도 (accuracy), 민감도 (sensitivity), 특이도 (specificity), 양성예측도 (Positive Predictive Value, PPV), 음성예측도 (Negative Predictive Value, NPV), AUROC (Area Under the Curve) 점수를 측정하였으며, 최종 결과 값은 10번의 예측을 수행한 결과에 대한 평균 결과로 산정되었다. 정성적[16] 평가 방법으로는 전립선암 악성도 예측 모델의 출력 벡터를 t-SNE(t-distributed Stochastic Neighbor Embedding)[17]로 시각화하여 분석하였다. 이 때, t-SNE 그림에 나타난 출력 벡터의 군집화 정도를 실루엣 점수(Silhouette score)[18]로 수치화하여 추가 제시함으로써 전립선암 악성도 예측 모델의 이진 분류 결과를 평가한다. 식 (3)의 실루엣 점수는 동일 클래스 내의 출력 벡터 밀집도와 서로 다른 클래스 간의 분리 정도를 측정하고 이를 -1과 1 사이의 값으로 정규화 하는 형태를 보이며, 1에 가까울수록 군집화가 잘 되었음을 의미한다.
이 때, α(i)는 출력 벡터 i가 속한 클래스 내의 다른 벡터들과의 거리 평균이며, b(i)는 출력 벡터 i가 속하지 않은 다른 클래스의 벡터들과의 거리 평균을 의미한다.
표 1은 ProstateX 데이터셋만을 사용해 자기지도학습 과정을 마친 모델과 SNUBHPCa 데이터셋으로 추가 학습을 수행한 모델을 바탕으로 단일 및 다중 파라미터 MR 영상에 대한 전립선암 악성도 예측 결과를 나타낸다.
실험 결과, 단일 파라미터 영상을 사용했을 때보다 다중 파라미터 영상들을 융합하여 전립선암 악성도를 예측한 경우 더 높은 성능을 보였다. SNUBPCa 데이터셋으로 자기지도학습을 추가 수행한 모델에서 단일 파라미터 MR 영상의 가장 좋은 성능을 보인 T2wMR 영상의 예측 성능 대비 다중 파라미터 MR 영상을 사용한 예측 융합 방식의 성능이 정확도, 특이도, 양성예측도, AUROC 점수에서 각각 5.97%, 15.59%, 11.76%, 0.03이 향상되었다. 이 때, 민감도에서 5.71%의 하락이 보이긴 하지만 특이도에서 큰 향상이 있어 전체 정확도까지 개선되는 효과를 보였다. 특히 예측 융합 방식 중에서는 과반수 투표 방식이 더욱 효과적이었는데, 이는 평균 투표 방식이 확률값 예측을 평균화하기 때문에 잘못된 판단의 확률이 최종 예측값에도 반영되지만, 과반수 투표 방식은 이진화된 예측값에서 과반수의 예측만으로 판단되기 때문에 전립선암 악성도 예측에 유리함을 의미한다. 따라서 전립선암 악성도 예측 시, 단일 파라미터 MR 영상만을 고려하기보다는 다중 파라미터 MR 영상 정보를 예측 융합 방식으로 모두 활용하는 것이 효과적이다.
또한 ProstateX 만으로 자기지도학습 과정을 완료한 모델보다 SNUBPCa로 자기지도학습 과정을 추가 수행한 모델이 단일 파라미터 MR 영상을 사용했을 때와 다중 파라미터 MR 영상을 사용했을 때 모두의 경우에서 더 높은 정확도를 보였다. 두 종류의 자기지도학습 모델 중 SNUBPCa로 자기지도학습을 추가로 수행한 모델이, 모든 단일 및 다중 파라미터 MR 영상의 실험 결과에 대해 정확도, 민감도, 음성예측도, AUROC 점수가 평균적으로 0.8%, 4.49%, 1.77%, 0.02가 향상되었다. 해당 실험 결과를 바탕으로, 전립선암 악성도 예측에 사용할 데이터와 유사한 특징을 가진 공공 데이터셋 뿐만 아니라 실제 전립선암 악성도 예측에 사용할 데이터셋으로 자기지도학습 과정을 추가 수행하면, 인코더가 전립선암 악성도 예측에 더 유용한 특징들을 추출하여 성능 향상에 도움이 됨을 알 수 있다.
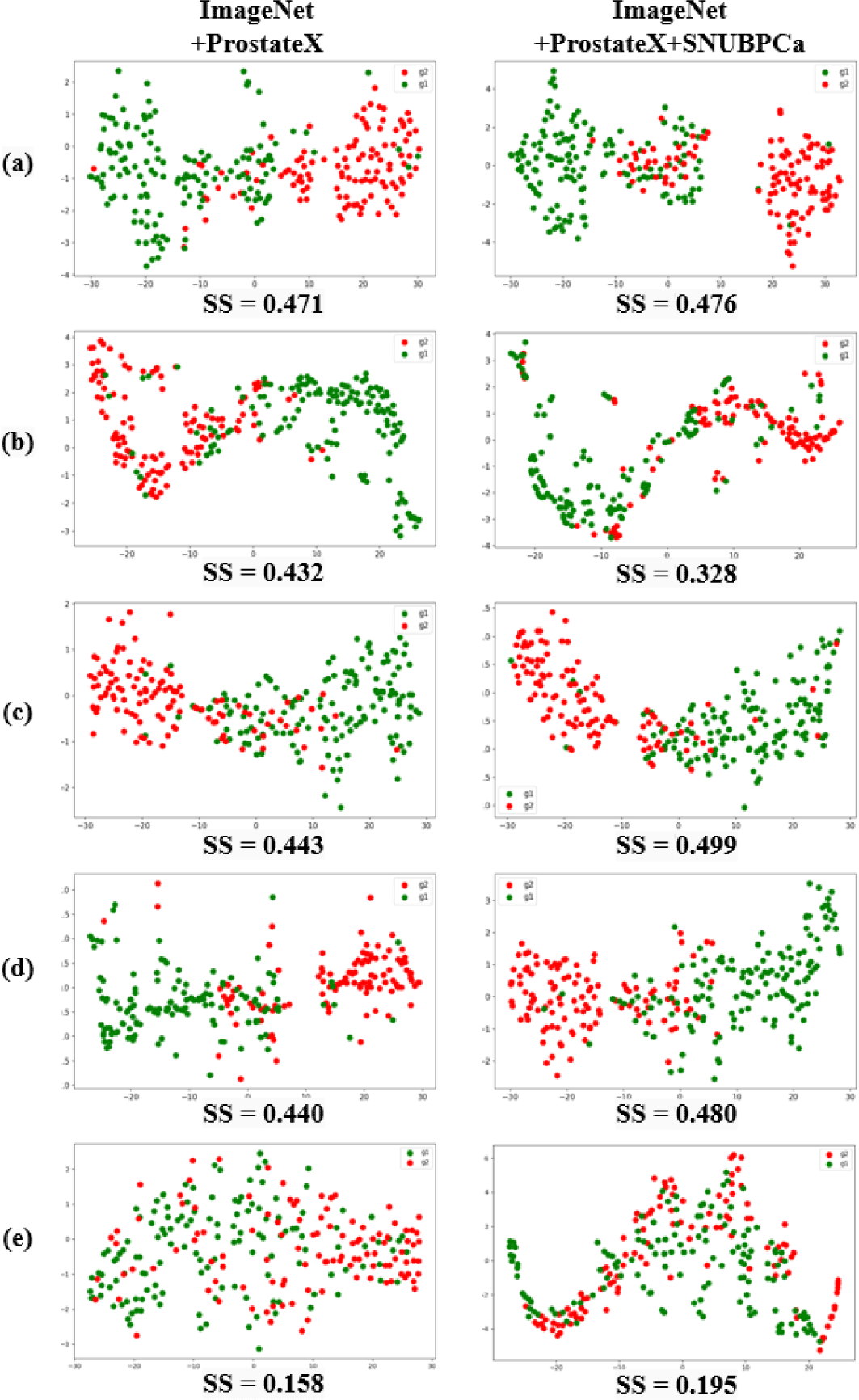
그림 4는 전립선암 악성도를 예측하기 위해 추가 학습을 수행하기 전과 후의 모델 출력 벡터를 시각화한 t-SNE 분석 그림과, 각 그림 당 출력 벡터들의 군집화 정도를 수치화한 실루엣 점수이다. 대부분의 결과에서 SNUBPCa로 자기지도학습을 추가로 수행한 모델이 추가 학습을 수행하지 않은 모델에 비해 고위험군과 저위험군의 특징 벡터 간 거리가 더 멀어지고 동일 클래스 내의 특징 벡터 간 거리는 줄어들었음을 확인할 수 있다. 단일 파라미터 MR 영상 중 T2wMR 영상을 사용한 경우, t-SNE 시각화 결과에서는 고위험군과 저위험군의 특징 벡터를 이진화한 결과 간에 간격이 생기고, 실루엣 점수는 0.005 점 향상한 점을 통해 자기지도학습을 추가 수행했을 때 서로 다른 클래스 간의 분포 구분이 뚜렷해짐을 확인할 수 있다. ADCmap의 경우는 동일 클래스 내의 특징 벡터 간 밀집도는 높아지고 서로 다른 클래스의 특징 벡터 간에는 거리가 멀어지면서 실루엣 점수가 0.056점 향상했는데, 이는 표 1에서 분류 정확도가 1.29% 향상된 것과 경향성이 일치한다. 또한 다중 파라미터 MR 영상을 사용하는 영상 융합 방식과 특징 융합 방식에서는 고르게 분산되어 있었던 특징 벡터의 분포가 추가 학습 이후 구조화된 패턴을 보이며 각 클래스 내 특징 벡터 분포의 일관성이 증가하였다. 이는 실루엣 점수 0.04, 0.37의 향상과 정확도 0.16%, 0.32%의 상승으로 나타나 두 그룹 간의 구분 성능이 개선되었음을 확인할 수 있다. 이를 통해 전립선암 악성도를 예측할 데이터셋을 자기지도학습 과정에 추가할 시 고위험군과 저위험군 각각의 주요 영상 정보를 명확하게 추출하여 모델의 예측 성능 향상에 기여함을 알 수 있다.
4. 결론
본 논문에서는 전립선암 악성도 예측을 위해 다중 파라미터 MR 영상들을 융합하고, 자기지도학습 모델을 전립선암 악성도 예측에 사용할 데이터로 추가 자기지도학습 하는 방법을 제안하였다. 실험 결과, 단일 파라미터 영상만을 사용했을 때보다 다중 파라미터의 영상을 예측 융합하여 전립선암 악성도를 예측했을 때 예측 성능이 향상되었으며, 예측 융합 방식 중에서도 과반수 투표 방식이 가장 효과적이었다. 또한 자기지도학습 네트워크에서 모델을 획득할 때, 전립선암 악성도를 예측할 영상을 사용해 추가 학습을 시키는 것이 대부분의 평가 지표에서 성능 향상을 가져왔으며, 특히 민감도에서 가장 큰 향상폭을 보였다. 이를 통해 의료 영상과 같이 데이터셋 및 레이블 데이터 확보에 제한적인 환경에서, 최종 전립선암 악성도를 예측할 데이터셋을 활용하여 자기지도학습 모델을 미세 조정하는 것이 예측 및 분류 성능을 향상시킬 수 있음을 보였다.




