1. 서론
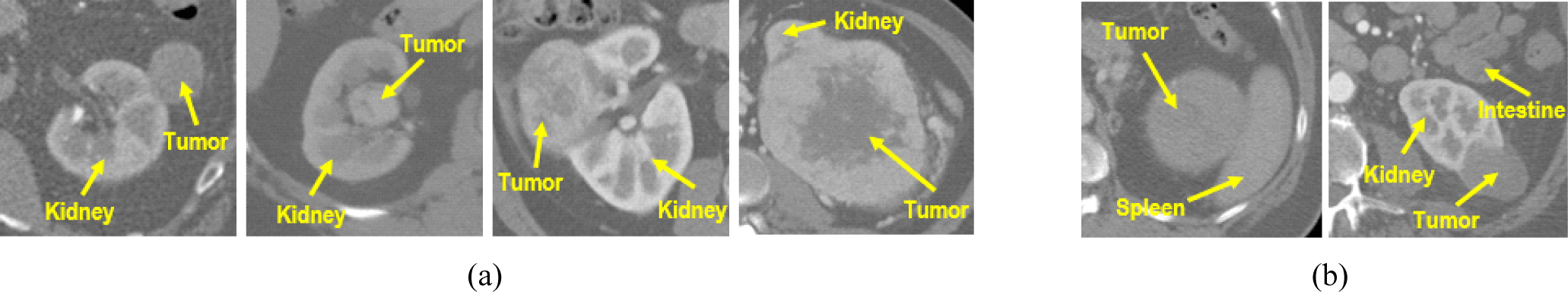
신장암은 전체 성인 종양 중 2~3%를 차지하며 전 세계적으로 43만건 이상의 신장 질환 사례가 발견되고 있다. 대부분 초기에 특별한 증상이 없어 증상이 나타났을 때 이미 질환이 악화된 경우가 많으므로 조기에 발견하여 수술적 치료를 하는 것이 중요하다[1-6]. 과거에는 신장과 신장 주위의 전이 조직이나 림프선을 절제하는 근치적신장절제술이 일반적이었지만, 최근에는 신장을 최대한 보존하면서 종양만을 절제하는 부분신장절제술이 더 많이 수행되고 있다[7]. 부분신장절제술을 계획하기 위해서는 신장 종양의 위치, 형태 및 수술 중 주요 조직 보호를 위한 안전 마진(safety margin)을 파악하는 것이 중요하므로 신장 종양을 분할하는 것이 필요하다. 그러나 그림 1과 같이, 크고 작은 다양한 크기의 종양이 환자의 신장 내부, 외부, 경계 등 여러 위치에 존재하며, 소장과 비장 같은 주변 장기와 형태 및 밝기값이 유사하여 해당 기관으로 과대 분할(over-segmentation)과 이상치(outlier)가 발생할 수 있으므로 신장 종양을 분할하는 데 한계가 있다.
복부 CT 영상에서 신장 종양을 분할하기 위한 관련 연구는 일반적으로 지도학습(supervised learning)을 통해 수행되며 2019 Kidney and kidney Tumor Segmentation (KiTS19) Challenge에서 제공된 공개 데이터를 사용하여 성능을 평가한다. 복부 3D CT 영상에서 신장 종양의 대략적인 위치를 확인하기 위해 위치 식별 및 분할 단계로 이루어진 계단식 V-Net[8]과 중요한 특징 정보를 추출하여 주요 부분에 중점을 두는 Squeeze and Excitation(SE) 모듈과 같은 어텐션을 활용한 U-Net[9,10]을 통해 54.16~74.32%의 신장 종양 분할 성능을 보였으나 작은 크기의 종양 검출에는 한계가 있었다. 또한, 자가적응형 프레임워크를 기반으로 하여 네트워크의 파라메터를 자동으로 최적화하는 nnU-Net에 잔차연결(residual connection)을 적용하거나, 저해상도 3D 영상에서 신장 영역을 추출한 후 고해상도 3D 영상으로 업샘플링한 뒤 신장과 신장 종양으로 각각 분할하거나 신장 위치화와 신장과 신장 종양을 분할하는데 활용하는 계단식 네트워크를 통해 신장 종양에 대해 82.54%~85.09%의 개선된 성능을 보였다[11,12,13]. 이와 같은 딥 컨볼루션 기반의 지도학습을 통한 신장 종양 분할의 경우 레이블 데이터가 부족한 경우 성능 저하가 발생할 수 있으므로 충분한 레이블 데이터를 확보하는 것이 중요하다. 그러나 의료 영상에서 레이블링에 소요되는 시간과 비용이 높아 레이블 데이터 수집이 어려운 한계가 있다[14-18]. 이러한 레이블 데이터 부족 문제를 완화하기 위해 레이블이 있는 데이터와 레이블이 없는 데이터를 함께 활용하여 효과적으로 훈련하는 준지도학습(semi-supervised learning) 방법이 필요하다. 준지도학습은 훈련 시 레이블이 없는 데이터의 정보를 추가적으로 활용하여 네트워크의 일반화 성능을 향상시켜 적은 레이블 데이터 세트에서 발생할 수 있는 과적합(overfitting) 문제를 완화한다.
준지도학습 분야에서는 불확실성을 가중치로 활용하는 손실함수를 적용한 평균-교사모델을 제안하여 교사 모델이 더 정확하고 낮은 불확실성을 가진 예측을 생성하도록 하는 신장 분할 연구와, 레이블이 있는 데이터에서 입력 영상과 레이블 간의 분포를 계산하고 이를 기반으로 레이블이 없는 데이터에 대한 신뢰성 있는 의사 레이블(pseudo label)을 생성하는 3D U-Net을 제안하여 훈련 초기에 레이블이 있는 데이터에 과적합 되는 문제를 해결하기 위한 신장 분할 연구가 진행되었다[19,20]. 이외에도 신경교종과 비인두암종과 같은 종양에 대해서는 모델에서 레이블이 없는 데이터에 대한 높은 품질의 의사 레이블을 생성하기 위해 레이블이 있는 데이터에 적대적 손실(adversarial loss)을 적용하여 훈련 안정성을 높이거나, 교사 모델의 예측으로부터 불확실성을 계산하여 패널티를 부여하는 손실함수와 전역적 문맥 정보를 파악하기 위한 셀프-어텐션 (Self-Attention) 모듈을 사용한 평균-교사 모델[21,22] 을 이용한 연구가 수행되었다. 그러나 신장 종양에 대한 연구 사례는 아직 없다.
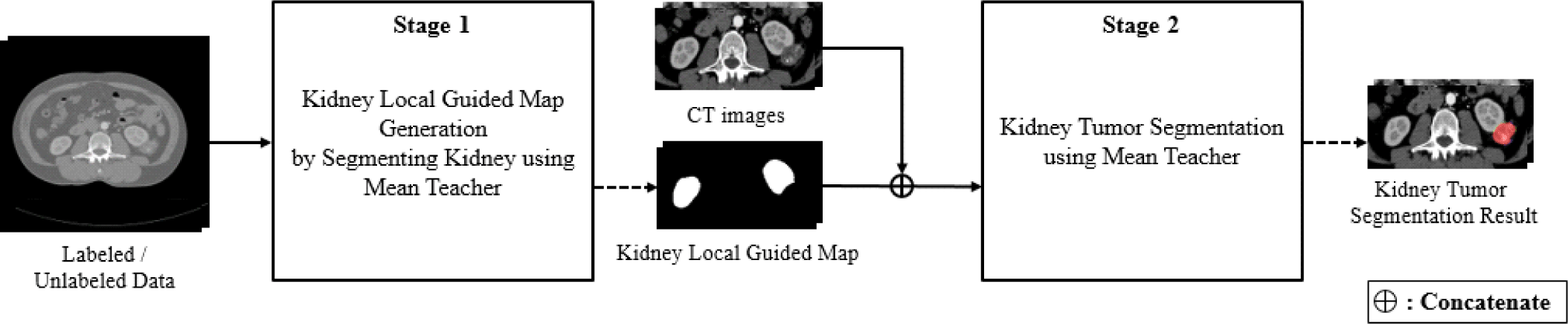
따라서 본 논문에서는 레이블 데이터가 부족한 상황에서 작은 크기의 신장 종양을 효과적으로 검출하는 것을 목표로, 레이블이 없는 데이터를 함께 활용하여 신장의 위치 정보를 담은 신장 로컬 가이드 맵을 생성하고 이를 추가 입력 채널로 활용하여 신장 주변의 종양에 집중하여 분할하는 평균-교사 네트워크를 제안한다. 또한, 기본 U-Net과 신장 및 신장 종양 분할에서 우수한 성능을 보이는 nnU-Net과의 성능 비교를 수행하며, 종양 크기에 따라 4개의 그룹으로 분류하여 결과를 분석한다.
2. 제안 방법
본 제안 방법은 그림 2와 같이 두 단계의 평균-교사 모델[23]로 구성되며, 신장의 위치 정보를 포함하는 신장 로컬 가이드 맵을 생성하는 신장 분할 단계와 신장 로컬 가이드 맵을 통해 신장 주변의 작은 크기 종양에 중점을 둔 신장 종양 분할단계로 이루어진다.
복부 CT 영상에서 신장이 상대적으로 작은 영역을 차지하기 때문에 신장의 지역적 문맥 정보에 집중하여 분할하기 위해 512x512 크기의 전체 복부 CT 영상을 사용하는 대신 신장 영역만을 포함하도록 영상을 자르고 이를 400x256 크기로 조정한다.
복부 CT 영상에서 신장 종양과 주변 조직의 밝기값이 유사한 경우, 다른 장기나 주변 조직을 신장 종양으로 판단하여 오분할이 발생할 수 있다. 따라서 그림 3과 같이 영상의 밝기값을 조정하여 신장 종양과 주변 조직이 잘 구별될 수 있도록 밝기값 정규화를 수행한다. 밝기값 정규화를 위해 식 (1)과 같이 영상의 밝기값을 최소-최대 정규화(min-max normalization)을 통해 0~255 사이로 정규화한다.
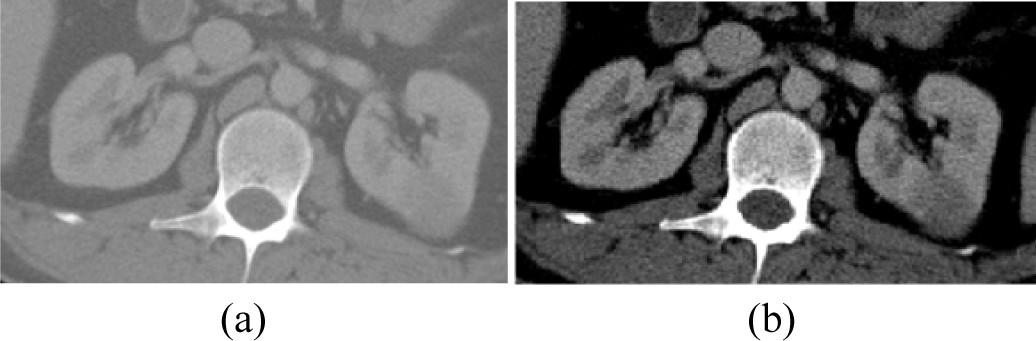
이 때, I는 원본 영상의 밝기값이고, Imin와 Imax은 각각 사용하고자 하는 밝기값 범위의 최소값과 최대값으로, 신장과 관련 없는 조직의 영향을 줄이기 위해 지방과 연조직 범위를 나타내는 –79HU와 304HU로 설정한다[11].
복부 CT 영상에서 신장 종양은 신장과 인접한 위치에서 발생하기 때문에 신장의 위치 정보를 활용하여 신장 종양의 위치를 파악할 수 있다. 먼저 평균-교사 모델을 사용하여 신장을 분할한 후, 분할 결과를 신장 로컬 가이드 맵으로 사용하여 종양 분할 단계에서 신장의 위치 정보를 제공한다.
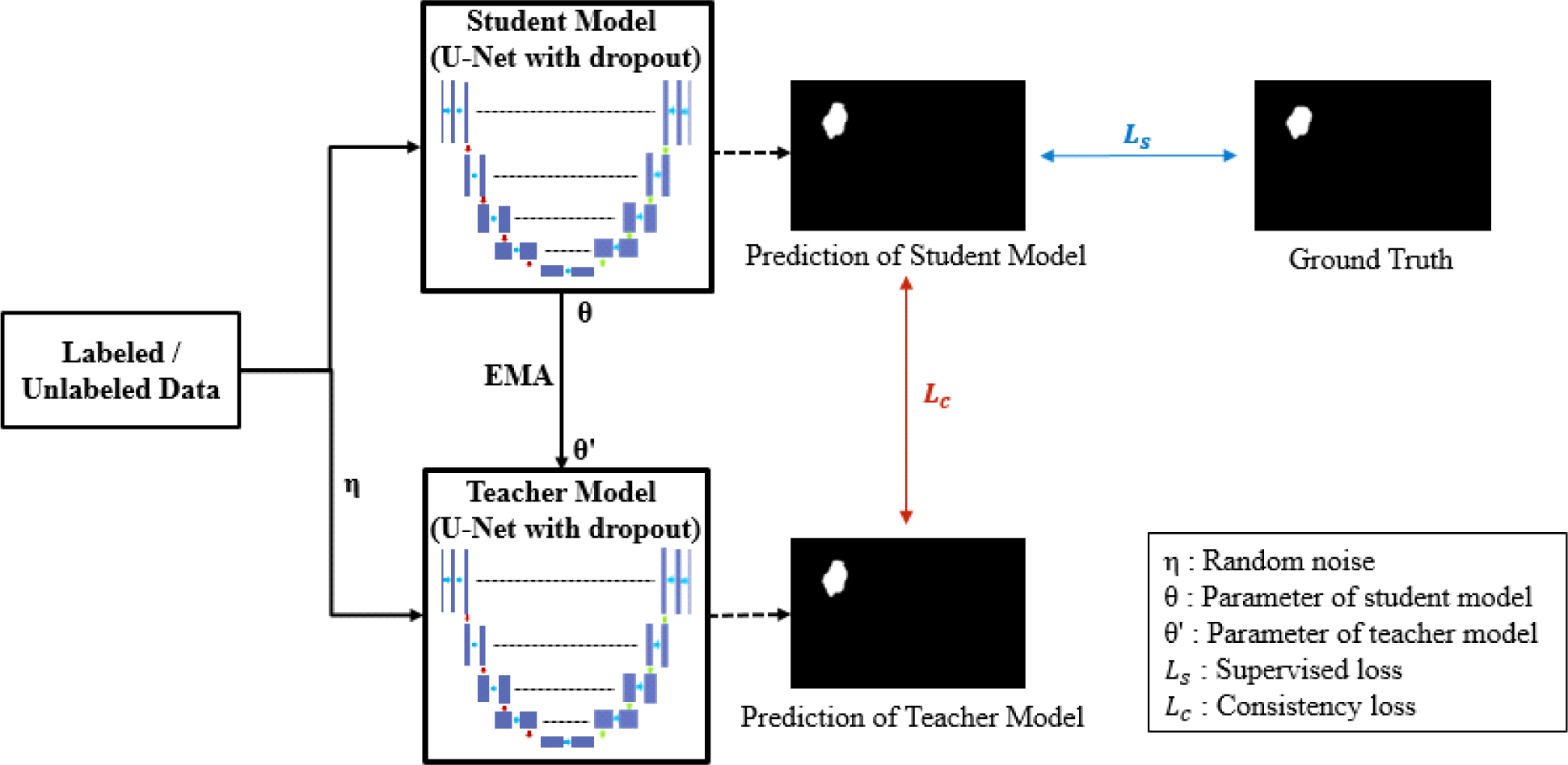
평균-교사 모델은 그림 4와 같이 학생과 교사 모델로 구성되며, 레이블이 있는 영상과 레이블이 없는 영상을 모두 사용하여 준지도학습을 수행한다. 각 모델은 2D U-Net[24]을 백본 네트워크로 사용하며, 5개의 레이어로 이루어진 수축경로(contracting path)와 확장경로(expanding path)를 가진다. 수축경로의 각 레이어에서는 3x3 컨볼루션 연산, 배치 정규화와 ReLU 활성화 함수를 적용하고, 2x2 맥스풀링 및 드롭아웃을 통해 특징맵을 생성한다. 확장경로의 각 레이어에서는 2x2 업샘플링을 거친 특징맵과 동일한 레이어에 위치한 수축경로의 특징맵을 연결연산(skip connection)을 통해 결합하고 3x3 컨볼루션 연산, 배치 정규화와 ReLU를 반복한다. 마지막 레이어는 예측을 위한 확률맵을 생성하기 위해 시그모이드 활성화 함수를 사용하는 1x1 컨볼루션 레이어로 구성된다. 네트워크의 일반화 성능 향상을 위한 추가적인 섭동(perturbation)을 가하기 위해 드롭아웃이 적용되며, 교사 모델의 경우 입력 영상에 랜덤 노이즈를 적용한다.
학생 모델은 경사하강법을 사용하여 파라미터가 갱신되어 훈련이 진행되고, 교사 모델의 파라미터는 식 (2)와 같이 모든 훈련 단계에서 학생 모델 파라미터와 이전 단계의 교사 모델 파라미터 간의 지수이동평균(exponential moving average)으로 갱신된다.
이 때, θt는 교사 모델의 파라미터이고 θt-1는 이전 단계의 교사 모델 파라미터이고 θs는 학생 모델의 파라미터이고 α는 이동평균을 조절하는 하이퍼파라미터 값이고 0.99로 산정하였다.
이후 CT 영상과 신장 로컬 가이드 맵을 결합하여 생성한 두 개의 채널로 이루어진 영상을 평균-교사 모델의 입력 영상으로 사용하여 최종적으로 신장 종양을 분할한다. 신장 로컬 가이드 맵을 추가 입력 채널로 사용함으로써 신장의 위치 정보를 고려하며, 이를 통해 신장 주변 영역에 집중하여 작은 크기의 종양의 위치를 파악하는 데 도움이 될 수 있다.
학습을 위한 전체 손실 함수로 식 (3)과 같이 지도 손실함수(Ls)와 일관성 손실함수(Lc)를 함께 사용하여 레이블이 있는 데이터와 없는 데이터를 학습에 함께 활용한다.
지도 손실함수는 레이블이 있는 데이터에 대한 학습을 수행하는 손실 함수로, 학생 모델이 출력한 예측값과 정답 레이블 간의 차이를 계산하여 모델이 예측한 결과가 실제 정답과 가까워지도록 모델을 학습시킨다. 지도 손실함수는 식 (4)과 같이 이항교차엔트로피(Binary cross entropy)와 다이스 손실(Dice loss)의 합으로 계산된다.
일관성 손실함수는 레이블이 있는 데이터와 레이블이 없는 데이터 모두에 대한 학습을 수행하는 손실 함수로, 학생 모델과 교사 모델이 출력한 예측값 간의 차이를 최소화하여 일관성을 유지하도록 모델을 학습시킨다. 일관성 손실함수는 식 (5)와 같이 평균제곱오차(Mean-squared error)를 사용하여 계산된다. 이러한 손실함수들을 통해 모델은 정답 레이블이 필요하지 않아 레이블이 없는 데이터에 대한 학습이 가능해진다.
이 때, yi는 i번째 데이터의 정답 레이블이고 은 i번째 데이터에 대한 학생 모델을 통해 출력된 예측값이고 는 i번째 데이터에 대한 교사 모델을 통해 출력된 예측값이고 N은 전체 데이터 수이다.
3. 실험 및 결과
실험에서 사용된 데이터는 Minnesota Medical Center에서 제공한 KiTS19 Challenge 데이터로, 부분신장절제술 혹은 근치적신장절제술을 받은 210명 환자의 수술 전 복부 CT 동맥기 영상이다. 이 영상의 해상도는 512x512이며, 슬라이스 장수는 19~1059장, 슬라이스 두께는 0.4375~1.041mm, 슬라이스 간격은 0.5~5mm이다.
실험 환경은 GeForce RTX 3090 그래픽 카드가 장착된 서버에서 파이썬 기반의 딥러닝 라이브러리인 Pytorch 1.10.1 버전을 사용하여 수행되었다. 네트워크 학습 시 에폭은 200, 학습률은 0.003, 배치 크기는 8로 설정하였으며 최적화를 위해 아담 옵티마이저를 사용하였다. 또한 학생과 교사 모델의 각 레이어에 [0.005, 0.1, 0.2, 0.3, 0.5]의 드롭아웃을 적용하였다. 실험은 홀드아웃(hold-out) 검증 방식을 사용하였으며, 훈련 데이터 169개와 테스트 데이터 41개로 나누고, 훈련데이터에서 레이블이 있는 데이터와 레이블이 없는 데이터의 비율을 조정하면서 실험을 진행했다.
본 제안 방법의 평가를 위해 2D U-Net[23]과 2D nnU-Net[25]을 사용한 지도학습 방법과 평균-교사 모델(Mean Teacher, MT)과 신장 로컬 가이드 맵을 추가하여 신장위치 정보를 고려한 준지도학습 제안 방법을 비교하였으며, 레이블이 있는 데이터의 비율을 조정하여 네트워크 훈련에 사용하는 레이블 데이터 수에 따른 성능을 분석하였다. 신장 종양 크기 별 분할 성능을 평가하기 위해 종양의 크기를 기준으로 네 그룹으로 나누었다. 종양 크기가 2.3cm 미만인 경우 작은 크기 종양, 2.3cm 이상 4.3cm 미만인 경우 중간 크기 종양, 4.3cm 이상 7.4cm 미만인 경우 큰 종양, 7.4cm 이상인 경우 매우 큰 종양으로 설정했다[25]. 각 그룹 별 종양 크기의 평균 및 표준편차는 작은 크기 종양은 1.8±0.2cm, 중간 크기 종양은 3.1±0.5cm, 큰 종양은 5.1±0.7cm, 매우 큰 종양은 10.5±2.1cm로 나타났다.
본 제안 방법의 성능을 평가하기 위해 정량적 평가와 정성적 평가를 수행하였으며 정량적 평가에는 F1-score, 재현율(Recall), 정밀도(Precision)를 성능지표로 하여 제안 방법과 비교 방법 간의 분할 성능을 평가하였다. 또한 복부 CT 영상에서 신장 종양 영역은 배경 영역에 비해 상대적으로 적은 영역을 차지하므로 신장 종양과 배경의 비율이 불균형하므로 신장 종양 영역에 대한 성능을 균등하게 고려하기 위해 신장 종양과 배경의 정확도를 각각 계산한 후 평균을 취하는 Balanced accuracy를 추가적으로 평가하였다.
표 1은 레이블 데이터 비율에 따른 지도학습 방법과 준지도학습 방법의 신장 종양 분할 성능지표 결과이다. 지도학습으로 수행된 방법의 경우, 레이블이 있는 데이터 100%를 사용한 방법들에 비해 레이블이 있는 데이터를 25%만 사용한 방법들의 F1-score, 재현율, 정밀도 수치가 레이블 데이터의 비율이 줄어들면서 감소함을 나타냈다. 레이블이 있는 데이터 25%와 레이블이 없는 데이터 75%를 사용한 준지도학습 방법의 F1-score, 재현율, 정밀도는 26.23%, 27.50%, 34.66%로, 훈련 초기에 적은 레이블 데이터로 인해 과적합이 발생하고 일반화 성능이 낮아져 레이블이 없는 데이터를 충분히 활용하지 못해 과소 분할이 발생하여 낮은 성능을 보였다. 레이블이 있는 데이터 25%를 사용하고 신장 로컬 가이드 맵을 적용한 제안 방법의 경우 F1-score, 재현율, 정밀도 수치는 75.24%, 72.96%, 86.57%로 신장 로컬 가이드 맵을 사용하여 신장의 위치 정보를 고려함으로써 종양의 위치 파악에 도움이 되어 다른 비교 방법에서 발생한 작은 크기의 신장 종양에 대한 과소 분할 문제를 개선하고 가장 우수한 결과를 보였다. 또한, 제안 방법의 Balanced accuracy 측정 결과는 86.41%로 레이블이 있는 데이터 100% 사용한 nnU-Net에 비해 7.46%p 높은 성능을 나타내어 신장 종양 분할 정확도가 가장 크게 향상되었음을 보였다.
| Methods | Network | Ratio(%) | Metrics(%) | ||||
|---|---|---|---|---|---|---|---|
| Labeled | Unlabeled | Balanced accuracy | F1-score | Recall | Precision | ||
| Supervised learning | U-Net[24] | 100 | 0 | 69.7±16.0 | 43.4±33.0 | 39.7±32.3 | 61.3±39.2 |
| 25 | 0 | 64.4±14.3 | 30.5±28.8 | 29.5±29.0 | 39.5±36.3 | ||
| nnU-Net[25] | 100 | 0 | 78.9±17.7 | 61.2±34.4 | 58.0±35.6 | 76.9±34.1 | |
| 25 | 0 | 68.7±18.5 | 41.6±38.6 | 37.8±37.1 | 59.7±44.7 | ||
| Semi supervised learning | MT[23] | 25 | 75 | 63.4±13.2 | 26.2±27.2 | 27.5±26.7 | 34.6±35.0 |
| MT with Kidney Local Guided Map (Ours) | 25 | 75 | 86.4±14.7 | 75.2±26.8 | 72.9±29.4 | 86.5±24.6 | |
표 2는 신장 종양 크기 별 분할 성능 결과를 확인하기 위한 표이다. U-Net, nnU-Net과 같은 지도학습 방법 및 신장 로컬 가이드 맵을 사용하지 않은 준지도학습 방법인 MT의 경우, 종양의 위치를 파악하지 못해 복부 CT 영상에서 작은 크기 및 중간 크기 종양에 과소 분할이 발생하여 전반적으로 낮은 분할 성능을 보였다. 반면에, 신장 로컬 가이드 맵을 사용한 제안 방법의 경우 신장의 위치 정보를 고려하여 종양의 위치 파악에 도움이 되어 작은 크기 및 중간 크기의 종양에 대한 분할 정확도를 개선시켰으며, 큰 종양에서는 레이블이 있는 데이터 100%를 사용한 nnU-Net과 유사한 성능을 보였다. 그러나 신장 로컬 가이드 맵은 신장의 위치 정보만을 제공하기 때문에 종양이 신장에 비해 매우 크거나 신장 없이 종양만 존재하는 슬라이스의 경우 위치 정보가 제한되어 매우 큰 종양에서 레이블이 있는 데이터 100%를 사용한 nnU-Net에 비해 종양에 과소분할 경향을 보여 분할 성능이 낮아졌다.
| Methods | Network | Ratio(%) | Balanced accuracy(%) | ||||
|---|---|---|---|---|---|---|---|
| Labeled | Unlabeled | Small (tumor<2.3) | Medium (2.3≤tumor<4.3) | Large (4.3≤tumor<7.4) | Extremely Large (7.4≤tumor) | ||
| Supervised learning | U-Net[24] | 100 | 0 | 57.7±12.7 | 62.3±10.5 | 74.7±13.9 | 84.8±12.4 |
| 25 | 0 | 54.9±12.1 | 58.2±10.0 | 68.6±14.4 | 76.9±8.7 | ||
| nnU-Net[25] | 100 | 0 | 62.7±14.4 | 70.3±14.2 | 87.8±13.5 | 94.9±3.8 | |
| 25 | 0 | 53.3±8.5 | 59.1±14.8 | 75.4±14.8 | 88.3±11.1 | ||
| Semi supervised learning | MT[23] | 25 | 75 | 56.4±10.9 | 60.6±12.0 | 63.1±12.8 | 74.0±10.1 |
| MT with Kidney Local Guided Map (Ours) | 25 | 75 | 88.74±16.0 | 88.9±14.6 | 88.2±13.4 | 78.5±11.1 | |
그림 5는 복부 CT 영상에서 레이블 데이터 비율에 따른 지도학습과 준지도학습 방법의 종양 크기 별 신장 종양 분할의 정성적 결과이다. 그림 5(a)는 레이블이 있는 데이터 100%에서 지도학습을 기반으로 수행한 U-Net의 결과이고, 그림 5(b)는 레이블이 있는 데이터 25%에서 지도학습을 기반으로 U-Net을 수행한 결과이고, 그림 5(c)는 레이블이 있는 데이터 100%에서 지도학습을 기반으로 nnU-Net을 수행한 결과이고, 그림 5(d)는 레이블이 있는 데이터 25%에서 지도학습을 기반으로 nnU-Net을 수행한 결과이고, 그림 5(e)는 레이블이 있는 데이터 25%와 레이블이 없는 데이터 75%를 함께 사용하는 준지도학습의 평균-교사 모델을 수행한 결과이고, 그림 5(f)는 레이블이 있는 데이터 25%와 레이블이 없는 데이터 75%를 함께 사용하고 신장 로컬 가이드 맵을 적용한 평균-교사 모델을 수행한 결과이다. 작은 크기 종양 분할 결과인 데이터 1~3의 경우, 작은 크기의 신장 종양으로 인해 종양의 위치 파악이 어려워 (a)~(e) 방법들은 적절한 분할을 수행하지 못했다. 그에 반해, 신장 로컬 가이드 맵을 사용한 (f) 방법은 종양 위치 파악에 도움을 주어 개선된 분할 결과를 보였다. 중간 크기의 종양 분할 결과인 데이터 4~6의 경우, 작은 크기의 신장 종양보다는 종양의 크기가 커져 (a)~(e) 방법들은 약간의 분할 개선을 보였지만 여전히 과소 분할 문제가 발생하였다. 반면에, 종양 위치 파악에 이점을 보이는 (f) 방법은 가장 좋은 분할 결과를 보였다. 큰 크기의 종양 분할 결과인 데이터 7과 데이터 8의 경우, 레이블이 있는 데이터 25%를 사용한 (b)와 (d) 방법들은 적은 레이블 데이터로 인해 과소 분할이 발생했으며, 레이블이 있는 데이터 100%를 사용한 (a)와 (c) 방법들은 더 많은 레이블 데이터를 활용하여 과소 분할이 개선되었다. 반면에, 레이블이 있는 데이터 25%를 사용하고 신장 로컬 가이드 맵을 사용하는 (f) 방법은 신장 주변 조직을 신장 종양으로 잘못 분할하는 과대분할 문제가 발생하지만 레이블 데이터를 적게 사용하면서도 비슷한 성능을 보여주었다. 매우 큰 크기의 종양 분할 결과인 데이터 9과 데이터 10의 경우, (f) 방법은 신장의 위치 정보만을 고려하기 때문에 매우 큰 종양에서는 과소 분할되는 경향을 보였다.
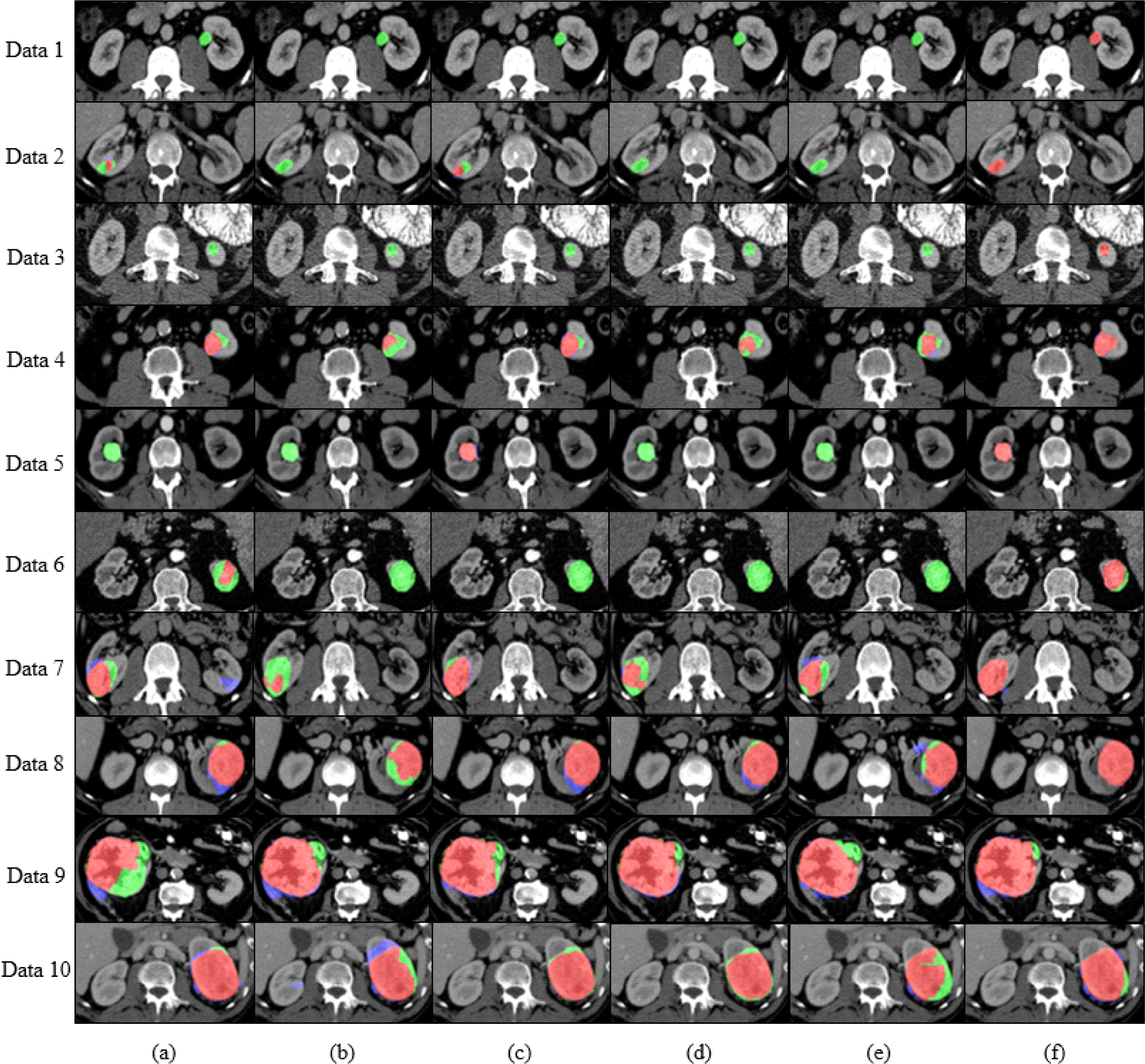
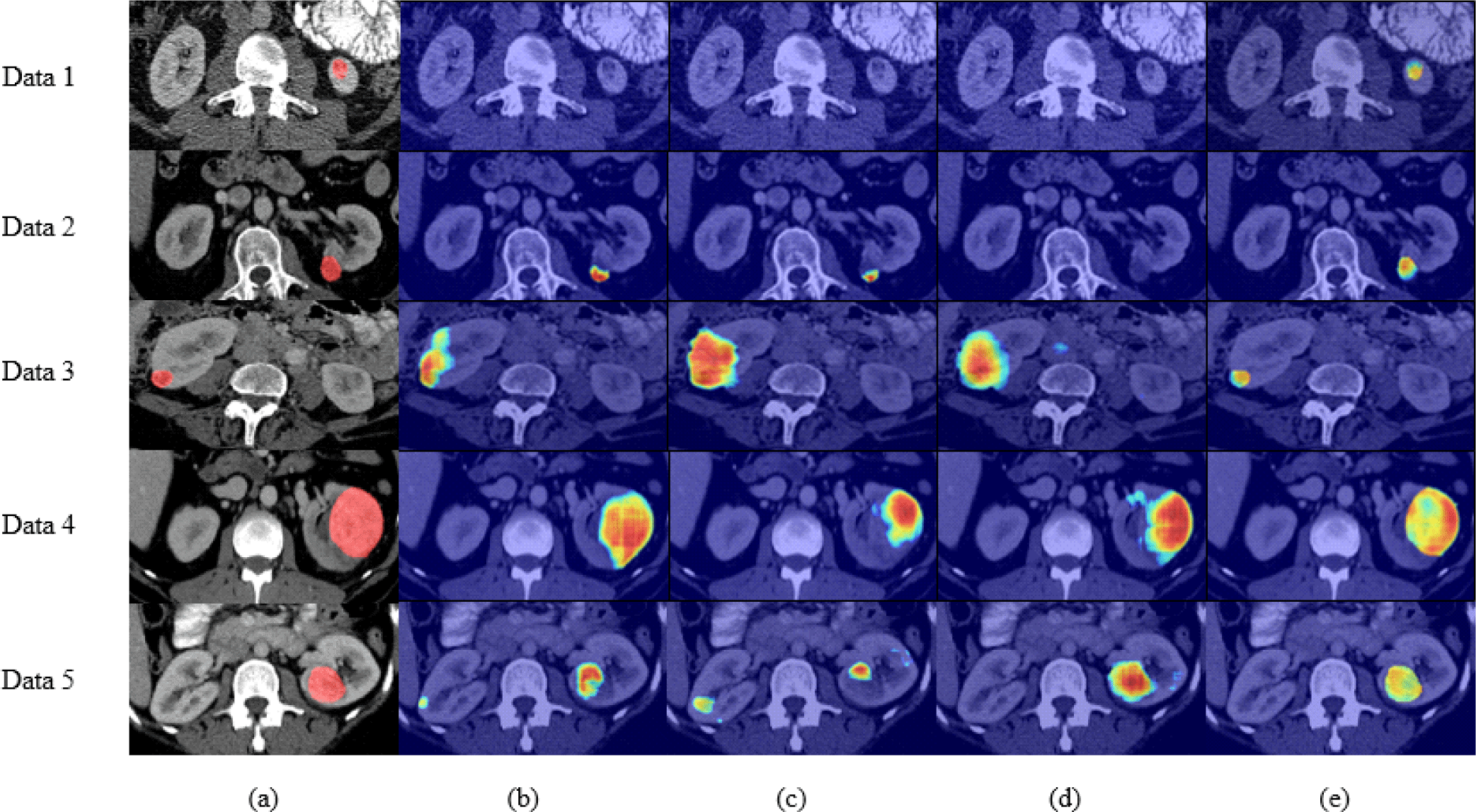
그림 6은 복부 CT 영상에서 레이블 데이터 비율에 따른 지도학습과 준지도학습 방법의 종양 크기별 신장 종양 분할의 Grad-CAM[27] 시각화 결과이다. 레이블이 있는 데이터 25%와 100%를 사용한 지도학습 방법의 경우, 신장 주위에 존재하는 작은 크기의 종양 위치를 제대로 파악하지 못하여 신장 종양의 일부에만 집중하거나 신장 영역까지 신장 종양 영역으로 분할하고 있는 것을 확인할 수 있다. 그러나 제안 방법은 신장의 위치 정보를 고려함으로써 적은 레이블 데이터 환경임에서도 다른 방법들에 비해 신장 종양 영역에 집중하여 분할하는 것을 확인할 수 있다.
4. 결론
본 논문에서는 복부 CT 영상에서 신장 주위의 다양한 위치에 나타나는 작은 크기의 신장 종양 분할을 위해 신장 위치 정보를 활용하는 신장 로컬 가이드 맵을 사용하는 준지도학습 방법인 평균-교사 네트워크를 제안하였다. 또한, 신장 종양의 크기에 따른 성능을 분석하였다. 작은 크기의 신장 종양은 복부 CT 영상 내에서 종양의 위치를 파악하기 어려워 분할 시 과소 분할이 일어나는 경향이 있으나, 신장 로컬 가이드 맵을 이용하는 제안 방법은 신장의 위치 정보를 고려하여 종양 주변에 집중함으로써 종양 위치 파악을 개선하였고, 특히 작은 크기의 종양에 대한 분할 성능을 향상시켰다. 그러나 신장 로컬 가이드 맵은 신장의 위치 정보만을 제공하므로 종양에 비해 신장의 크기가 매우 작거나 종양만 존재하는 슬라이스의 경우에는 고려할 수 있는 신장 위치 정보가 부족해 성능에 제한이 있다. 향후 연구로는 종양의 전역적 문맥 정보를 고려하여 크기가 큰 신장 종양에 대한 분할 성능을 개선하고자 한다.