Article
부분 신장 절제술 계획을 위한 하이브리드 CNN-트랜스포머 네트워크를 활용한 신장 종양 분할
김고운1,O
,
안진서1,O
,
이유빈1
,
홍헬렌1,*
Kidney Tumor Segmentation Using a Hybrid CNN-Transformer Network for Partial Nephrectomy Planning
Goun Kim1,O
,
Jinseo An1,O
,
Yubeen Lee1
,
Helen Hong1,*
Author Information & Copyright ▼
1Dept. of Software Convergence, Seoul Women’s University, Seoul, Republic of Korea
O These authors contributed equally to this work
© Copyright 2024 Korea Computer Graphics Society. This is an Open-Access article distributed under the terms of the
Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/4.0/) which permits
unrestricted non-commercial use, distribution, and reproduction in any
medium, provided the original work is properly cited.
Received: Jun 05, 2024; Revised: Jul 16, 2024; Accepted: Jul 30, 2024
Published Online: Sep 01, 2024
요약
신장암 치료를 위한 부분 신장 절제술에서 수술 계획을 위한 신장 종양의 정확한 크기와 위치 등의 정보가 필수적이다. 따라서 신장 종양을 정확하게 분할하는 것이 중요하지만, 종양이 주변 장기와 밝기값이 유사하고 위치 및 크기가 환자마다 다양하여 분할에 어려움이 있다. 본 연구에서는 신장 종양의 분할 성능 개선을 목표로, 영상 내 지역적 및 전역적 특징을 모두 고려할 수 있는 합성곱 신경망과 트랜스포머가 결합된 하이브리드 네트워크를 제안한다. 제안 방법은 UNETR++와의 비교 실험에서 다이스 유사계수 78.54%, 정밀도 85.07%로 전반적으로 우수한 성능을 보였다. 또한, 종양 크기별 분석에서는 UNETR++에서 관찰되는 과대 분할 및 이상치가 개선된 결과를 보였다.
Abstract
In partial nephrectomy for kidney cancer treatment, accurate segmentation of the kidney tumor is crucial for surgical planning, as it provides essential information on the precise size and location of the tumor. However, it is challenging due to the tumor’s similar intensity to surrounding organs and the variability in its location and size across patients. In this study, we propose a hybrid network that integrates a convolutional neural network and a transformer to capture both local and global features, aiming to improve the segmentation performance of kidney tumors. We validated our method through comparative experiments with UNETR++, outperforming it with a Dice Similarity Coefficient (DSC) of 78.54% and a precision of 85.07%. Moreover, in the analysis by tumor size, our method demonstrated improvements by reducing over-segmentation and outlier cases observed in UNETR++.
Keywords: 신장 종양 분할; CT 영상; 하이브리드 네트워크; 합성곱 신경망; 트랜스포머
Keywords: Kidney Tumor Segmentation; CT Image; Hybrid Network; Convolutional Neural Network; Transformer
1. 서론
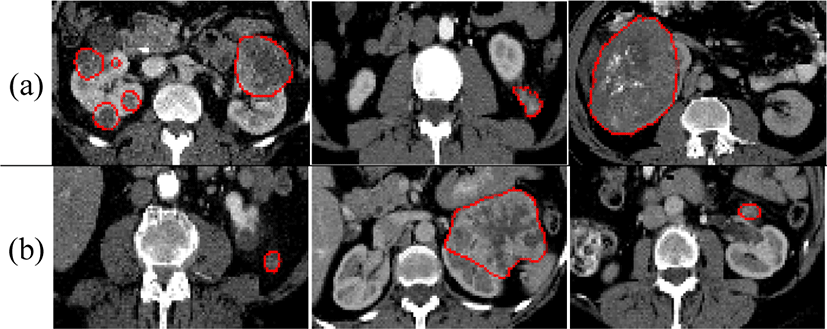
신장암은 신장에서 발생하는 악성 종양으로, 과거에는 신장 전체를 제거하는 근치적 신장 절제술이 일반적이었지만, 최근에는 종양 부위만 제거하는 부분 신장 절제술이 널리 사용되고 있다[1]. 수술 계획을 세우기 위해서는 신장 종양의 크기와 위치 등을 파악하는 것이 필수적이므로, 신장 종양의 정확한 분할이 중요하다. 그러나 그림 1(a)와 같이 환자마다 종양의 위치와 크기가 다양하며, 그림 1(b)와 같이 주변 장기와 밝기값이 유사하여 신장 종양을 분할하는 데 어려움이 있다.
Figure 1.
The challenging issue of kidney tumor segmentation: (a) the various locations and sizes of kidney tumors, (b) the similar intensity values between kidney tumors and surrounding organs. The red line represents the kidney tumor.
Download Original Figure
최근 의료영상에서 인체 구조물이나 질환 부위를 분할하는 연구에서는 합성곱 신경망(convolutional neural network)과 트랜스포머(transformer)를 주로 활용하고 있다. 신장 종양 분할을 위한 합성곱 신경망 기반 접근법에는 U-Net[2] 구조에 어텐션 매커니즘(attention mechanism)을 추가하는 방법, U-Net의 인코더 및 디코더 구조를 개선하는 방법 등이 있다. 어텐션 메커니즘을 추가하는 방법으로는, 스킵 연결(skip connection)에 어텐션 메커니즘을 더하거나[3, 4], 종양의 에지 정보를 강조하기 위해 어텐션 기반 디코더를 추가하거나[5], 인코더의 잔여 블록(residual block) 기반 컨볼루션 연산에 어텐션을 추가하는 방법[6]이 있다. U-Net의 인코더 구조를 개선하는 방식으로는 슬라이스 간 공간 상관관계 정보를 반영하기 위해 컨볼루션 연산과 LSTM을 결합한 ConvLSTM을 사용하는 방법[7]이 있다. 디코더 구조를 개선하는 방식으로는 디코더의 모든 레이어에서 다중 스케일 확률맵을 각각 출력하여 학습 손실 계산에 반영하는 방법[8]이 있다. 이러한 합성곱 신경망 기반 신장 종양 분할의 경우, KiTS19 챌린지(2019 Kidney and Kidney Tumor Segmentation Challenge) 데이터셋에서 74.32~91.1%의 다이스 유사계수(DSC, Dice Similarity Coefficient)를 보였다. 컨볼루션 연산을 통해 영상의 지역적 공간 맥락(local spatial context)을 잘 반영할 수 있지만, 전역적인 특징을 충분히 고려하지 못하므로 환자마다 다양한 위치와 형태의 신장 종양 분할에 한계가 있다.
전역적인 문맥 정보를 고려하는 트랜스포머 기반 접근 방법은 트랜스포머 블록으로 구성된 U자 모양의 인코더-디코더 구조를 주로 사용하며, 인코더나 디코더 내부의 트랜스포머 블록을 수정하거나 스킵 연결 및 병목 구간(bottleneck)을 개선하는 방법이 활용된다. 인코더와 디코더의 트랜스포머 블록을 수정하는 방법으로는 복부 CT 영상에서 장기를 분할하기 위해 이동(shifted) 윈도우 기반의 어텐션 메커니즘을 활용한 Swin Transformer 블록을 적용하여, 기존 트랜스포머의 멀티 헤드 어텐션(multi-head attention) 연산의 계산 복잡도를 줄이면서도 79.13%의 다이스 유사계수 성능을 보였다[9]. 트랜스포머의 어텐션 연산에 라플라시안 필터를 적용하여 에지(edge)나 텍스처(texture)와 같은 지역적 특징 정보를 추가로 활용한 방법은 81.09%의 다이스 유사계수 성능을 보였다[10]. 정확한 세포핵(nuclei) 분할을 위해 세포핵과 에지 간 상관관계를 활용하는 디코더를 별도로 구성하여 분할하는 방법은 97.01%의 다이스 유사계수 성능을 보였다[11]. 스킵 연결 및 병목 구간을 개선하는 방법으로는, COVID-19 감염 부위(infected region) 분할을 위해 흉부 X-선 영상 및 CT 영상의 인코더 특징맵을 병목 구간에서 트랜스포머 기반 크로스 어텐션(cross attention)으로 결합한 방법이 69.22%의 다이스 유사계수 성능을 보였다[12]. 그러나 트랜스포머 기반 방법은 전역적인 정보를 효과적으로 모델링할 수 있지만, 지역적인 정보 활용에는 한계가 있다. 이로 인해 영상 내 공간적으로 인접한 픽셀 간의 높은 상관관계가 간과될 수 있어 다양한 크기를 갖는 종양의 특징을 적절히 반영하기 어렵다는 문제점이 있다.
따라서 본 연구에서는 복부 CT 영상에서 종양 분할 성능 개선을 목표로, 합성곱 신경망과 트랜스포머를 결합한 하이브리드 CNN-Transformer 네트워크를 제안한다. 본 논문의 독창성은 다음과 같다.
-
다양한 위치와 크기의 신장 종양 분할을 위해 합성곱 신경망으로 추출한 특징맵을 여러 해상도를 고려하는 트랜스포머 기반 UNETR++[13, 14] 네트워크에 입력함으로써 전역 및 지역적 특징을 통합적으로 활용한다.
-
합성곱 신경망 연산 시 확장된 컨볼루션과 깊이별 분리 컨볼루션 블록(dilated and depthwise separable convolution)을 사용하여 종양의 특징을 효과적으로 추출한다.
-
트랜스포머 기반의 UNETR++를 비교 방법으로 하여 하이브리드 네트워크인 제안 방법과 균형 정확도(balanced accuracy), 다이스 유사계수, 정밀도(precision), 재현율(recall) 측면에서 비교하며, 신장 종양의 크기별로 분할 성능을 분석한다.
2. 제안 방법
그림 2는 본 제안 방법의 구조도를 나타낸다. 먼저, 복부 CT 영상에 대해 전처리 과정을 수행한 후, 확장 및 깊이별 분리 컨볼루션을 통해 특징맵을 추출한다. 다음으로, 추출된 특징맵을 트랜스포머 기반 UNETR++ 네트워크에 입력하여 최종적으로 분할 결과를 출력한다.
2.1 데이터 전처리
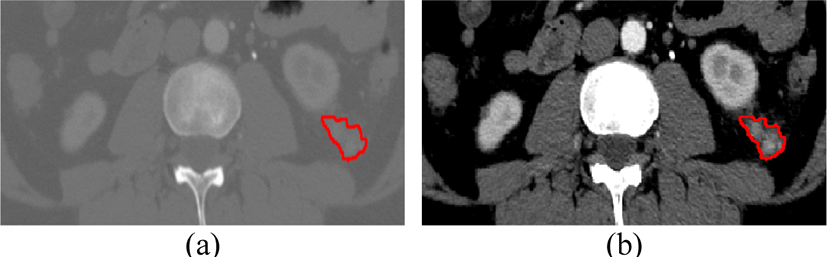
영상 전처리 단계에서는 신장의 지역적 정보에 집중하기 위해 복부 CT 영상을 신장 중심으로 자르고, 400x256 크기로 조정한다. 또한, 신장 종양은 주변 조직과 밝기값이 유사하여 구분이 어려우므로, 종양과 유사한 밝기값을 가지는 지방 및 연조직을 포함하는 [-79HU, 304HU] 범위로 최소-최대 정규화(min-max normalization)하여 0~255 사이로 조정함으로써 구분을 향상시킨다[15, 16].
Figure 3.
Data preprocessing example: (a) original CT image, (b) image after preprocessing. The red line represents the kidney tumor.
Download Original Figure
2.2 트랜스포머 기반 네트워크: UNETR++
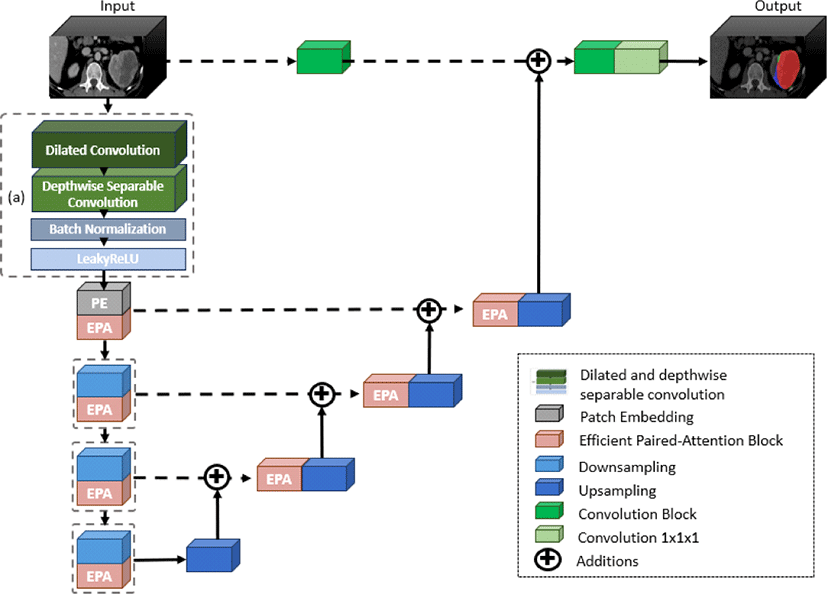
복부 CT 영상에서 신장 종양의 위치와 크기가 다양하기 때문에 영상 전체에 대한 전역적인 정보를 활용하는 것이 필요하다. 본 연구에서는 이를 고려한 트랜스포머 기반 네트워크인 UNETR++를 기반 네트워크로 사용한다. 일반적인 트랜스포머 기반 네트워크는 단일 해상도만 고려하고, 멀티 헤드 어텐션을 활용해 연산량이 증가하는 한계를 가진다. 반면 UNETR++는 다양한 해상도의 특징맵을 활용해 다양한 크기의 신장 종양을 효과적으로 분할하며, 계산 효율을 높이기 위해 EPA (Efficient Paired-Attention) 블록을 사용한다.
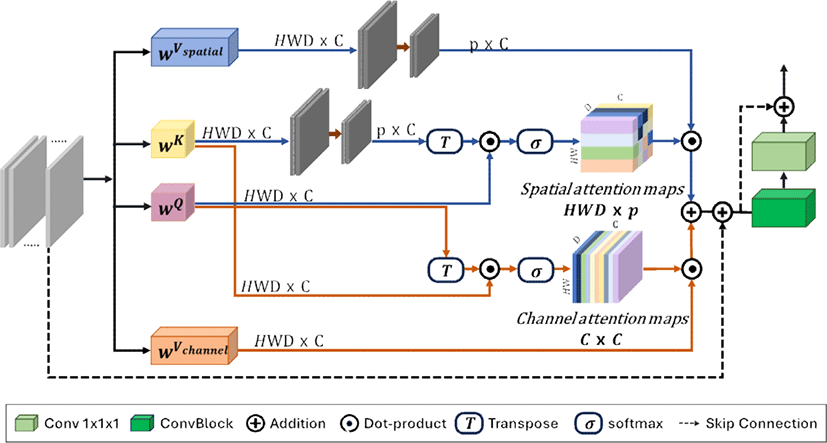
UNETR++는 U자 구조를 가지며, 네 단계의 인코더(encoder)와 디코더(decoder)로 구성되어 있고, 각 단계는 EPA 블록으로 구성된다. 인코더의 첫 번째 단계에서는 패치 임베딩을 통해 입력 영상을 3D 패치로 나누고 EPA 블록을 수행한다. 나머지 세 단계는 다운샘플링(downsampling)과 EPA 블록으로 구성된다. 디코더는 처음 세 단계에서 업샘플링(upsampling)을 수행하고, 스킵 연결을 통해 인코더로부터 추출된 특징맵을 전달받은 후 EPA 블록을 수행한다. 디코더의 마지막 단계에서는 업샘플링 후 입력 영상과의 스킵 연결을 수행한다. 마지막으로, 디코더에서 얻은 특징맵에 컨볼루션을 적용하여 최종적으로 신장 종양 분할 결과를 출력한다. EPA 블록은 그림 4와 같이 공간 어텐션(spatial attention)과 채널 어텐션(channel attention)으로 구성되며, 이를 통해 공간 정보와 채널 특징맵 간의 상호 의존성을 효과적으로 학습할 수 있다. 각 어텐션 모듈은 쿼리(query), 키(key), 값(value)을 사용하여 연산을 수행하며, 두 어텐션 모듈 모두 쿼리와 키를 공유함으로써 모델 복잡도를 줄이는 효과를 갖는다.
Figure 4.
The structure of the EPA block. The blue and orange lines represent spatial and channel attention, respectively.
Download Original Figure
2.3 지역적 특징 추출을 위한 컨볼루션 블록
트랜스포머 기반 네트워크는 전역적인 정보 활용에 효과적이지만 신장 종양의 세부적인 크기 및 형태 특징 추출을 위한 지역적인 정보 활용에 한계가 있다. 따라서 본 연구에서는 지역적인 특성을 고려하는 합성곱 신경망을 추가하는 하이브리드 방식을 제안한다. 합성곱 신경망에서는 확장 및 깊이별 분리 컨볼루션을 사용하는데, 이는 그림 2(a)와 같이 확장된(dilated) 컨볼루션, 깊이별 분리(depthwise separable) 컨볼루션, 배치 정규화, 그리고 LeakyReLU 활성화 함수를 순차적으로 수행한다.
확장된 컨볼루션은 확장률(dilation rate)에 따라 컨볼루션 필터 내부에 제로 패딩(zero padding)을 추가하여 수행되는데, 이 방법은 추가적인 매개변수 없이 넓은 수용 영역(receptive field)을 제공하여 더 넓은 컨텍스트 정보를 효과적으로 추출할 수 있다. 신장 종양의 분할 성능을 향상시키기 위해, 3x3x3 크기 커널에 대해 확장률을 2로 설정하여 5x5x5 컨볼루션을 적용한 효과를 얻는다.
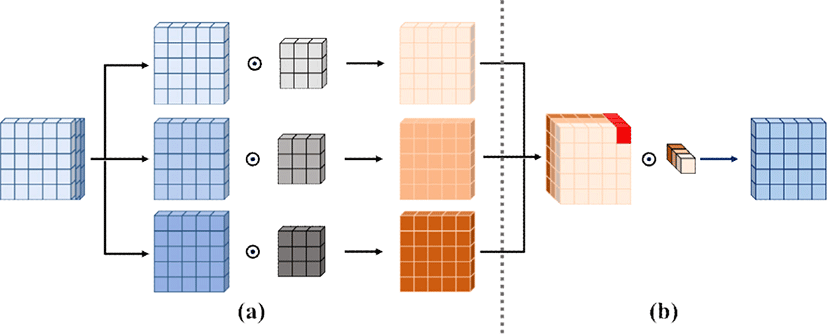
깊이별 분리 컨볼루션은 깊이별 컨볼루션과 점별(pointwise) 컨볼루션을 분리하여 순차적으로 수행하는 방법으로, 매개변수 수와 계산량을 줄여 연산의 효율성을 높일 수 있다. 깊이별 컨볼루션은 각 채널에 대해 서로 다른 필터를 사용하여 독립적으로 연산을 수행한 후 채널들을 결합(concatenate)하는데, 이는 그림 5(a)와 같이 수행된다. 그 후 그림 5(b)의 점별 컨볼루션은 1x1xC 크기의 컨볼루션 필터를 사용하여 각 픽셀에 대한 모든 채널의 정보를 통합하고 하나의 채널로 출력한다. 이때 C는 깊이별 컨볼루션에서의 출력 채널 수를 나타낸다.
Figure 5.
The process of dilated and depthwise separable convolution: (a) depthwise convolution, (b) pointwise convolution.
Download Original Figure
3. 실험 및 결과
실험에서 사용한 데이터는 KiTS19 챌린지에서 제공한 공개 데이터[17, 18]로, 2010년부터 2018년까지 미네소타 대학 의료 센터에서 신장 절제술을 받은 환자 중 무작위로 선택된 210명의 복부 CT 영상이다. CT 영상은 19~1059장의 슬라이스로 구성되며, 해상도는 512x512, 슬라이스 두께는 0.4375mm~1.041mm, 슬라이스 간격은 0.5mm~5mm이다.
실험은 NVIDIA GeForce RTX 3090 GPU, Intel Xeon CPU, 32GB RAM이 장착된 Ubuntu 20.04.6 LTS 운영체제의 서버에서 수행하였고, CUDA 11.4 버전의 GPU 환경에서 파이썬(python) 3.8 및 파이토치(pytorch) 1.12.1 라이브러리를 활용하였다. 네트워크를 학습하기 위해 최적화 함수로 확률적 경사 하강법(Stochastic Gradient Descent, momentum=0.95, weight_decay=3e-5)과 손실 함수로는 교차 엔트로피(cross entropy)를 사용하였다. 학습률(learning rate)은 0.001, 배치 크기(batch size)는 2 또는 4, 에폭(epoch)은 교차검증 시 데이터셋 폴드에 따라 750~1700 사이의 값으로 설정하였다.
본 연구에서는 제안 방법의 성능을 평가하기 위해 정량적 및 정성적 분석을 수행하였다. 기반 네트워크인 UNETR++와의 성능을 비교하였으며, 특히 신장 종양의 크기별 효과에 대해 소형(small), 중형(medium), 대형(large), 극대형(extremely large) 네 개의 그룹으로 나누어 분석하였다[19]. 추가적으로, KiTS19 데이터셋을 사용한 기존 연구 결과와 비교 분석하였다. 정량적 평가에는 균형 정확도, 다이스 유사계수, 정밀도, 재현율을 측정하여 비교 방법과의 분할 성능을 평가하였다. 정성적 평가에서는 네트워크 분할 결과와 정답 레이블을 오버레이하여 시각적으로 다양한 위치와 크기의 신장 종양에 대한 분할 성능을 비교 분석하였다. 제안 방법의 네트워크 학습 및 평가를 위해 전체 210개 데이터에 대해 종양 크기별 데이터 개수의 비율을 고려한 계층별 k-폴드 교차 검증(stratified k-fold cross validation) 방식을 수행한다. 표 1은 교차 검증을 위한 각 폴드의 데이터 구성을 나타낸다.
Table 1.
Composition of the K-Fold dataset
| Tumor size |
Fold1 |
Fold2 |
Fold3 |
Fold4 |
Fold5 |
| Small |
8 |
8 |
8 |
8 |
8 |
| Medium |
16 |
16 |
15 |
15 |
13 |
| Large |
13 |
12 |
12 |
12 |
11 |
| Extremely large |
6 |
6 |
7 |
7 |
9 |
| Total |
43 |
42 |
42 |
42 |
41 |
Download Excel Table
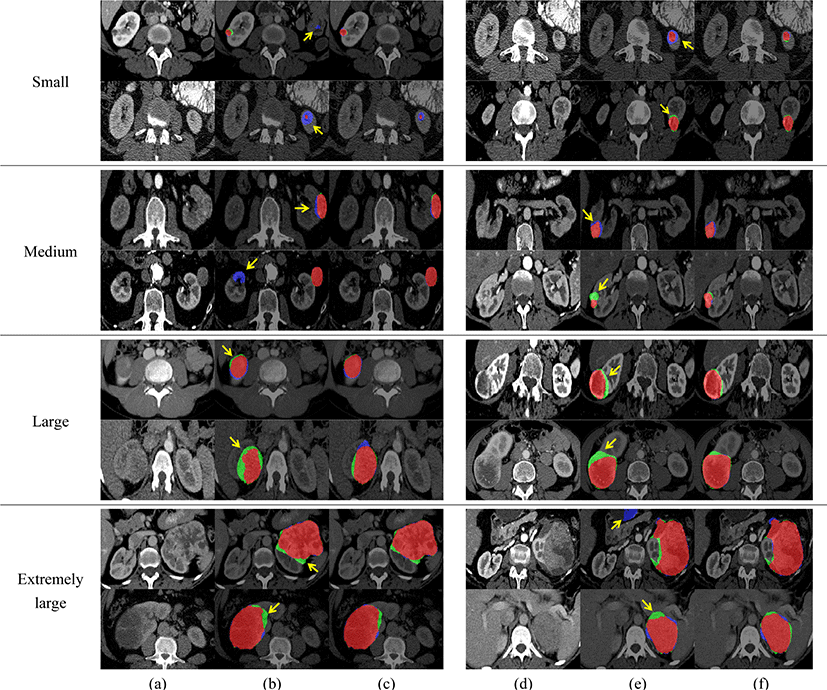
표 2는 신장 종양 분할에 대한 종양 크기별 및 전체적인 정량적 성능평가 결과를 나타낸다. 제안 방법은 대형 및 극대형 종양에서 UNETR++ 보다 모든 측면에서 성능이 향상되었으며, 소형 및 중형 종양에 대해서는 과대 분할이 개선되어 정밀도 측면에서 개선되는 결과를 보였다. 전반적으로, 제안 방법은 다이스 유사계수 및 정밀도가 각각 78.54%, 85.07%로 개선된 성능을 보였다. 이러한 결과는 그림 6의 정성적 성능평가 결과와도 일치한다. 대형 및 극대형 종양의 경우 제안 방법이 과소 및 과대 분할 문제를 개선하는 것으로 나타났으며, 소형 및 중형 종양에서는 과대 분할 및 이상치(outlier)가 개선되는 것을 보였다.
Table 2.
Performance evaluation of kidney tumor segmentation by size
| Size |
Methods |
Balanced Accuracy(%) |
DSC (%) |
Precision (%) |
Recall (%) |
| Small (tumor<2.3cm) |
UNETR++ |
81.92±16.72
|
57.32±31.89
|
69.93±29.35 |
63.90±33.44
|
| Ours |
80.09±19.72 |
56.37±37.84 |
71.82±27.78
|
60.20±39.43 |
| Medium (2.3cm≤tumor<4.3cm) |
UNETR++ |
88.98±12.39
|
77.23±24.25
|
83.57±19.61 |
78.04±24.80
|
| Ours |
88.73±12.65 |
77.14±23.45 |
85.87±16.34
|
77.53±25.34 |
| Large (4.3cm≤tumor<7.4cm) |
UNETR++ |
94.46±4.88 |
86.45±10.34 |
86.34±13.43 |
89.23±9.78 |
| Ours |
94.65±4.98
|
87.05±9.87
|
87.04±13.16
|
89.53±10.00
|
| Extremely large (7.4cm≤tumor ) |
UNETR++ |
96.28±3.28 |
91.85±6.91 |
91.71±8.72 |
93.09±6.56 |
| Ours |
96.52±2.85
|
92.29±7.54
|
92.18±9.18
|
93.54±5.56
|
| Overall |
UNETR++ |
90.43±11.81
|
78.48±24.08 |
83.13±20.71 |
81.08±23.69
|
| Ours |
90.07±12.95 |
78.54±25.42
|
85.07±18.92
|
80.33±25.97 |
Download Excel Table
Figure 6.
Results of kidney tumor segmentation in abdominal CT images classified by size. (a) and (d) CT image, (b) and (e) segmentation results using UNETR++, (c) and (f) segmentation results using proposed method. The red, green, blue colors represent true positive, false negative(under-segmentation), false positive(over-segmentation), respectively. The yellow arrows in (b) and (e) indicate false negative and false positive regions.
Download Original Figure
표 3은 KiTS19 데이터셋을 활용한 기존 신장 종양 분할 연구와 제안 방법의 성능을 비교한 결과를 나타낸다. 관련연구 [4-8]의 경우 학습, 검증 및 테스트 데이터의 구성 비율이 본 연구와 차이가 있어 직접적인 성능 비교에는 한계가 있었다. 합성곱 신경망 기반 방법은 지역적 특징을 효과적으로 활용하는 반면, 트랜스포머 기반 네트워크는 전역 정보와 장거리 의존성을 효과적으로 모델링하여 다양한 위치와 크기의 신장 종양 분할에서 더 높은 다이스 유사계수를 보였다. 제안 방법은 합성곱 신경망과 트랜스포머의 장점을 결합하여 78.54%의 우수한 다이스 유사계수 성능을 달성하였다.
Table 3.
Comparison of the proposed method with previous studies using the KiTS19 dataset
| Methods |
DSC (%) |
| CNN |
Guo et al. [4] |
77.0 |
| Myronenko et al. [5] |
81.03 |
| XiuzhenXie et al. [6] |
74.32 |
| Kang et al. [7] |
78.91 |
| Zhao et al. [8] |
80.5 |
| Transformer |
UNETR++ |
78.48 |
| Hybrid |
Ours |
78.54 |
Download Excel Table
4. 결론
본 연구에서는 복부 CT 영상에서 다양한 크기 및 형태를 갖는 신장 종양의 분할 성능 향상을 위해 트랜스포머 기반 UNETR++을 기반 네트워크로 하여 합성곱 신경망 기반의 확장 및 깊이별 분리 컨볼루션 블록을 결합한 하이브리드 네트워크를 제안하였다. 제안 방법 성능을 UNETR++와 비교 분석한 결과, 소형 및 중형 종양에서는 과대 분할 및 이상치가 개선되었고, 대형 및 극대형 종양에서는 모든 성능 지표가 향상되어 전체적으로는 다이스 유사계수 78.54%, 정밀도 85.07%의 우수한 결과를 보였다. 제안 방법은 합성곱 신경망과 트랜스포머를 통해 전역 및 지역적 특징을 통합적으로 활용하였으며, 모든 크기의 종양 분할 성능을 개선하는 데 효과적이었다.
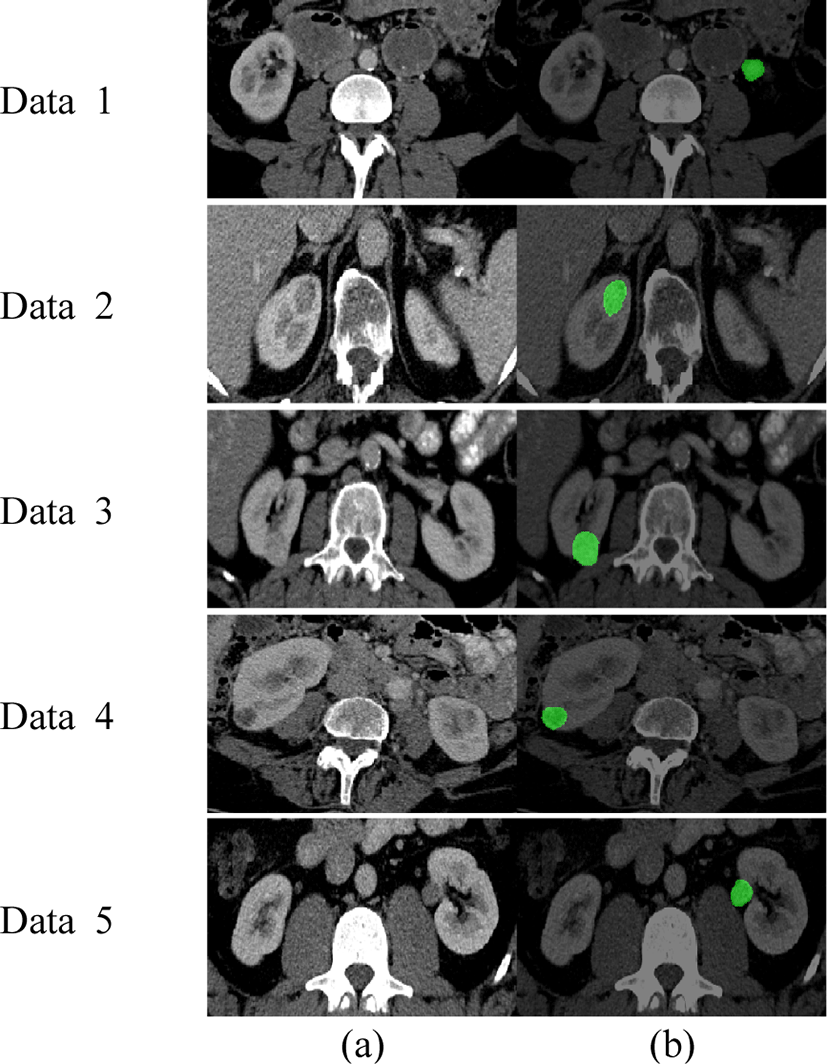
그러나 소형 종양은 넓은 수용 영역의 컨볼루션 연산 수행 시 검출(detect)되지 않는 한계가 있어, 그림 7과 같이 과소 분할이 나타났으며, 이로 인해 비교적 낮은 성능을 보였다. 따라서 향후 연구에서는 합성곱 신경망과 트랜스포머 블록을 효과적으로 결합하여 인코더의 구조를 개선함으로써 종양 크기의 변화에도 견고하게 대응할 수 있는 분할 성능을 목표로 한다.
감사의 글
본 연구는 보건복지부의 재원으로 한국보건산업진흥원의 보건의료기술연구개발사업 지원(HI22C1496) 및 서울여자대학교 학술연구비의 지원(2024-0218)을 받아 수행되었으며 이에 감사드립니다.
References
M.M. Picken, W. Lu, and N.G. Gopal, “Positive Surgical Margins in Renal Cell Carcinoma: Translating Tumor Biology Into Clinical Outcomes,”
Amercian Journal of Clinical Pathology, 143(5), pp. 620-622, 2015.



O. Ronneberger, P. Fischer, and T. Brox, “U-Net: Convolutional Networks for Biomedical Image Segmentation,”
Medical Image Computing and Computer Assisted Intervention (MICCAI) 2015, pp. 234-241, 2015.


P. Sun, Z. Mo, F. Hu, F. Liu, T. Mo, Y. Zhang, et al., “Kidney Tumor Segmentation Based on FR2PAttU-Net Model,”
Frontiers in Oncology, 12, pp. 1-13, 2022.




J. Guo, W. Zeng, S. Yu, and J. Xiao, “RAU-Net: U-Net Model Based on Residual and Attention for Kidney and Kidney Tumor Segmentation,”
IEEE International Conference on Consumer Electronics and Computer Engineering (ICCECE), pp. 353-356, 2021.


A. Myronenko, and A. Hatamizadeh, “3D Kidneys and Kidney Tumor Semantic Segmentation using Boundary-Aware Networks,”
arXiv:1909.06684, 2019.


X. Xie, L. Li, S. Lian, S. Chen, and Z. Luo, “SERU: A cascaded SE-ResNeXT U-Net for kidney and tumor segmentation,”
Concurrency and Computation: Practice and Experience, 32(14), pp. 1-11, 2020.


L. Kang, Z. Zhou, J. Huang, and W. Han, “Renal tumors segmentation in abdomen CT Images using 3D-CNN and ConvLSTM,”
Biomedical Signal Processing and Control, 72, pp. 1-16, 2022.


W. Zhao, D. Jiang, J.-P. Queralta, and T. Westerlund, “MSS U-Net: 3D segmentation of kidneys and tumors from CT images with a multi-scale supervised U-Net,”
Informatics in Medicine Unlocked, 19, pp. 1-11, 2020.


H. Cao, Y. Wang, J. Chen, D. Jiang, X. Zhang, Q. Tian, et al., “Swin-Unet: Unet-Like Pure Transformer for Medical Image Segmentation,”
European Conference on Computer Vision (ECCV) 2022, pp.205-218, 2022.


R. Azad, A. Kazerouni, B. Azad, E. Khodapanah Aghdam, Y. Velichko, U. Bagci, et al., “Laplacian-Former: Overcoming the Limitations of Vision Transformers in Local Texture Detection,”
Medical Image Computing and Computer Assisted Intervention (MICCAI) 2023, pp. 736-746, 2023.




Z. He, M. Unberath, J. Ke, and Y. Shen, “TransNuSeg: A Lightweight Multi-task Transformer for Nuclei Segmentation,”
Medical Image Computing and Computer Assisted Intervention (MICCAI) 2023, pp. 206-215, 2023.


Q. Guan, Y. Xie, B. Yang, J. Zhang, Z. Liao, Q. Wu, et al., “Unpaired Cross-Modal Interaction Learning for COVID-19 Segmentation on Limited CT Images,”
Medical Image Computing and Computer Assisted Intervention (MICCAI) 2023, pp. 603-613, 2023.


A.M. Shaker, M. Maaz, H. Rasheed, S. Khan, M.H. Yang, and F.S. Khan, “UNETR++: Delving into Efficient and Accurate 3D Medical Image Segmentation,”
IEEE Transactions on Medical Imaging, pp. 1-14, 2024.



F. Isensee, K.H. Maier-Hein, “An attempt at beating the 3D U-Net,”
arXiv:1908.02182, 2019.


N. Heller, F. Isensee, K.H. Maier-Hein, X. Hou, C. Xie, F. Li, et al., “The state of the art in kidney and kidney tumor segmentation in contrast-enhanced CT imaging: Results of the KiTS19 challenge,”
Medical Image Analysis, 67, pp. 1-16, 2021.




N. Heller, N. Sathianathen, A. Kalapara, E. Walczak, K. Moore, H. Kaluzniak, et al., “The KiTS19 Challenge Data: 300 Kidney Tumor Cases with Clinical Context, CT Semantic Segmentations, and Surgical Outcomes,”
arXiv:1904.00445, 2019.

E. Yang, C.K. Kim, Y. Guan, B.-B. Koo, and J.-H. Kim, “3D multi-scale residual fully convolutional neural network for segmentation of extremely large-sized kidney tumor,”
Computer Methods and Programs in Biomedicine, 215, pp. 1-12, 2022.



< 저 자 소 개 >
김 고 운

-
2023년 8월 서울여자대학교 소프트웨어융합학과 졸업(학사)
-
2023년 9월~현재 서울여자대학교 컴퓨터학과 소프트웨어융합전공 석·박사통합과정
-
관심분야 : 의료 인공지능, 딥러닝, 영상분할, 영상처리
안 진 서

-
2021년 8월 서울여자대학교 소프트웨어융합학과 졸업(학사)
-
2022년 3월~현재 서울여자대학교 컴퓨터학과 소프트웨어융합전공 석·박사통합과정
-
관심분야 : 의료 인공지능, 딥러닝, 영상분할, 영상처리
이 유 빈

-
2024년 2월 서울여자대학교 소프트웨어융합학과 졸업(학사)
-
2024년 3월~현재 성균관대학교 인공지능융합학과 석·박사통합과정
-
관심분야 : 멀티모달딥러닝, 사용자데이터분석, 영상처리
홍 헬 렌

-
1994년 2월 이화여자대학교 전자계산학과 졸업(학사)
-
1996년 2월 이화여자대학교 전자계산학과 졸업(석사)
-
2001년 8월 이화여자대학교 컴퓨터학과 졸업(박사)
-
2001년 9월~2003년 7월 서울대학교 컴퓨터공학부 BK 조교수
-
2006년 3월~현재 서울여자대학교 소프트웨어융합학과 교수
-
관심분야 : 의료 인공지능, 딥러닝, 영상처리 및 분석